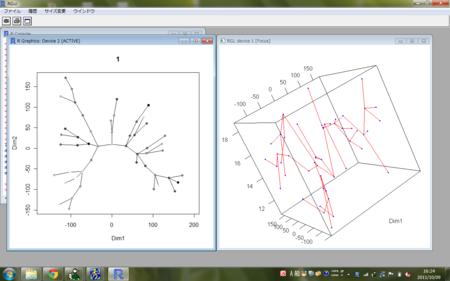
- 木(グラフ)があって、そのノードに値があるとする
- グラフは2次元にプロットできる
- ノードが持つ値を第3軸に与えて3次元プロットしてみよう
library(rgl)
library(vegan)
Ns<-50
Nm<-40
X<-matrix(sample(0:2,Ns*Nm,replace=TRUE),Ns,Nm)
D<-dist(X,method="manhattan")
MSTOri<-spantree(D)
AF<-apply(X,2,mean)
MAF<-AF
MAF[which(MAF>0.5)]<-1-MAF[which(MAF>0.5)]
SAF<-sign(AF-0.5)
R<-exp(-MAF)*SAF
ks<-1
for(i in 1:length(ks)){
P<-X%*%R
P<-P*ks[i]
Y<-cbind(P,X)
tmpD<-dist(Y,method="manhattan")
MST<-spantree(tmpD)
meanP<-mean(P)
tmpPlot<-plot(MST,col=gray((max(P)-P)/(max(P)-min(P))),cex=1,pch=19,main=ks[i])
plot3d(cbind(tmpPlot[[1]],P),col=4)
First<-2:(length(MST[[1]])+1)
Second<-MST[[1]]
tmpX<-c(t(cbind(tmpPlot[[1]][First,1],tmpPlot[[1]][Second,1])))
tmpY<-c(t(cbind(tmpPlot[[1]][First,2],tmpPlot[[1]][Second,2])))
tmpZ<-c(t(cbind(P[First],P[Second])))
segments3d(tmpX,tmpY,tmpZ,col=2)
}
