- 今、M次元格子上にN個の点を取る
- M次元格子は(0,1,2)としてみる
- これらの点はその遠近関係(以下のソースではマンハッタン距離にしてある)により、最小全域木を取ることができる
- 今、このN個の点にある値を付与しよう
- この値は相対的なものであるとすると、定数倍しても意味は変わらない
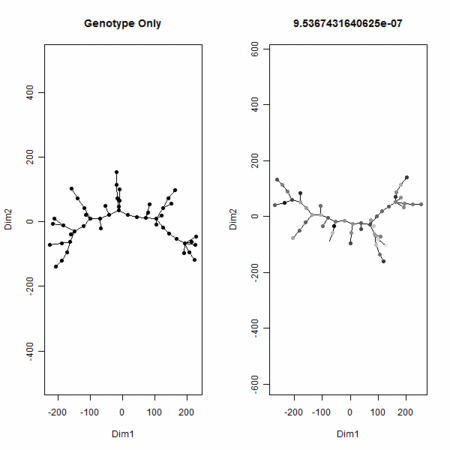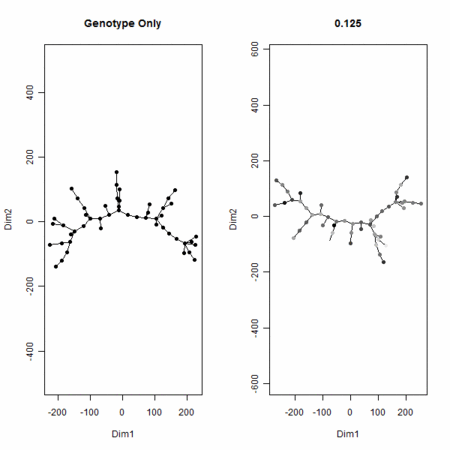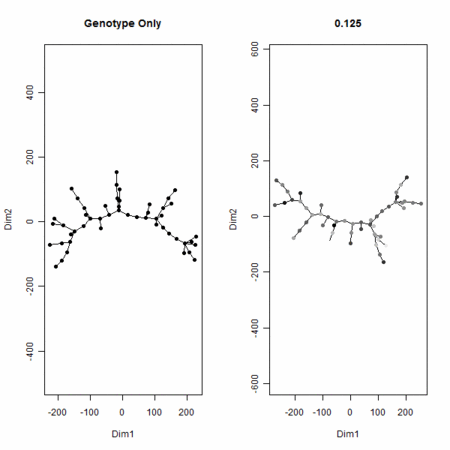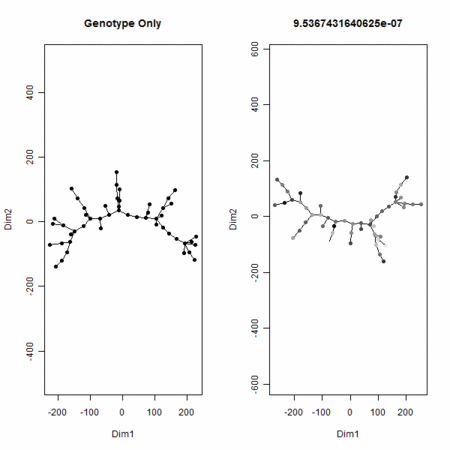
- しかしながら、この付与した値をも考慮してN個の点の座標をM+1次元空間の点と取りなおして最小全域木を撮りなおせば、付与した値のスケールが大きくなると、その値の多寡の影響力が大きくなり、付与した値のスケールが小さくなると、その影響がなくなる(木のトポロジーは、元のM次元情報で確定する)
- こちらで使っているMSTの話とつながる話(極言すれば、MSTはジェノタイプだけで決めて、そのうえで、各点にもう1次元分の値を付与することを考えてもよい。そのときに1次元分を付与して変化するMSTの辺の長さの和は(特にマンハッタン距離でやっていれば)、簡単に書き下せる形になる。
library(vegan)
library(animation)
Ns<-50
Nm<-40
X<-matrix(sample(0:2,Ns*Nm,replace=TRUE),Ns,Nm)
D<-dist(X,method="manhattan")
saveGIF({
MSTOri<-spantree(D)
AF<-apply(X,2,mean)
MAF<-AF
MAF[which(MAF>0.5)]<-1-MAF[which(MAF>0.5)]
SAF<-sign(AF-0.5)
R<-exp(-MAF)*SAF
ks<-2^(c(seq(from=-20,to=5,length=26),c(seq(from=5,to=-20,length=26))))
for(i in 1:length(ks)){
P<-X%*%R
P<-P*ks[i]
Y<-cbind(P,X)
tmpD<-dist(Y,method="manhattan")
MST<-spantree(tmpD)
meanP<-mean(P)
par(mfcol=c(1,2))
plot(MSTOri,cex=1,pch=19,main="Genotype Only")
plot(MST,col=gray((max(P)-P)/(max(P)-min(P))),cex=1,pch=19,main=ks[i])
}
}
, interval = 1, movie.name = "bm_demo.gif", ani.width = 600,
ani.height = 600)
