- 3次元の位置を色識別するために、ルールを入れて色塗りしてみる
my.hsv <- function(z,int0=0.6,sat0=0.3,int1=1,sat1=1){
arg <- Arg(z)
s <- which(arg<0)
arg[s] <- arg[s]+2*pi
r <- Mod(z)
s <- which(r>1)
r[s] <- log(r[s])
r. <- 4*(r%%1)
k <- floor(r.)
r. <- r.-k
inten <- sat <- rep(0,length(r))
s <- which(k==0)
inten[s] <- int1
sat[s] <- sat1-(sat1-sat0)*r.[s]
s <- which(k==1)
inten[s] <- int1-(int1-int0)*r.[s]
sat[s] <- sat0
s <- which(k==2)
inten[s] <- int0
sat[s] <- sat1-(sat1-sat0)*(1-r.[s])
s <- which(k==3)
inten[s] <- int1-(int1-int0)*(1-r.[s])
sat[s] <- sat1
return(cbind(arg,inten,sat))
}
my.hsv2rgb <- function(h,s,v){
hi <- floor(h/(2*pi)*6)
hi[which(hi==6)] <- 0
f <- (h/(2*pi)*6) %%1
p <- v*(1-s)
q <- v *(1-f*s)
t <- v *(1-(1-f)*s)
r <- g <- b <- rep(0,length(h))
s <- which(hi==0)
r[s] <- v[s];g[s] <- t[s]; b[s] = p[s];
s <- which(hi==1)
r[s] <- q[s];g[s] <- v[s]; b[s] = p[s];
s <- which(hi==2)
r[s] <- p[s];g[s] <- v[s]; b[s] = t[s];
s <- which(hi==3)
r[s] <- p[s];g[s] <- q[s]; b[s] = v[s];
s <- which(hi==4)
r[s] <- t[s];g[s] <- p[s]; b[s] = v[s];
s <- which(hi==5)
r[s] <- v[s];g[s] <- p[s]; b[s] = q[s];
return(cbind(r,g,b))
}
my.3d.angles <- function(x){
x. <- x/sqrt(apply(x^2,1,sum))
theta <- acos(x.[,3])
phi <- Arg(x.[,1]+1i*x.[,2])
return(cbind(phi,theta))
}
my.angle.complex.color <- function(angles){
w <- angles[,1] + 1i * angles[,2]
hsv <- my.hsv(w,int0=0.1,sat0=0.1,int1=1,sat1=1)
col <- my.hsv2rgb(hsv[,1],hsv[,3],hsv[,2])
return(rgb(col[,1],col[,2],col[,3]))
}
x <- seq(from=-4,to=4,len=100)
xx <- expand.grid(x,x)
z <- xx[,1]+1i * xx[,2]
my.f <- function(z){
(z^2-1)*(z-2-1i)^2/(z^2+2+2*1i)
}
w <- my.f(z)
hsv <- my.hsv(w,int0=0.1,sat0=0.1,int1=1,sat1=1)
col <- my.hsv2rgb(hsv[,1],hsv[,3],hsv[,2])
plot(xx,pch=20,col=rgb(col[,1],col[,2],col[,3]))
my.random.walk <- function(n.step,d,step=c(-1,0,1),prob=c(0.2,0.6,0.2)){
r <- matrix(sample(step,n.step*d,replace=TRUE,prob=prob),ncol=d)
return(apply(r,2,cumsum))
}
my.cell.walk.dt <- function(nt,n.iter,n.step,d=3,step.t = c(-2,-1,0,1,2),prob.t=c(0.1,0.2,0.4,0.2,0.1),step=c(-1,0,1),prob=c(0.2,0.6,0.2)){
ctr <- my.random.walk(nt,d=d,step=step.t,prob=prob.t)
ret <- matrix(0,0,d+1)
for(i in 1:nt){
tmp <- matrix(0,0,d)
for(j in 1:n.iter){
tmp2 <- my.random.walk(n.step,d=d,step=step,prob=prob)
tmp <- rbind(tmp,tmp2)
tmp <- unique(tmp)
}
tmp3 <- t(t(tmp) + ctr[i,])
tmp4 <- cbind(tmp3,rep(i,length(tmp3[,1])))
ret <- rbind(ret,tmp4)
}
return(ret)
}
my.cell.walk.dt.multi <- function(n.cell,nt,n.iter,n.step,d=3,step.t = c(-2,-1,0,1,2),prob.t=c(0.1,0.2,0.4,0.2,0.1),step=c(-1,0,1),prob=c(0.2,0.6,0.2),xyz=c(100,100,100)){
ret <- matrix(0,0,d+1)
for(i in 1:n.cell){
tmp <- my.cell.walk.dt(nt,n.iter,n.step,d,step.t,prob.t,step,prob)
x <- sample(1:xyz[1],1)
y <- sample(1:xyz[2],1)
z <- sample(1:xyz[3],1)
tmp <- t(t(tmp) + c(x,y,z,0))
ret <- rbind(ret,tmp)
}
return(ret)
}
n.cell <- 1
nt <- 2
n.iter <- 50
n.step <- 20
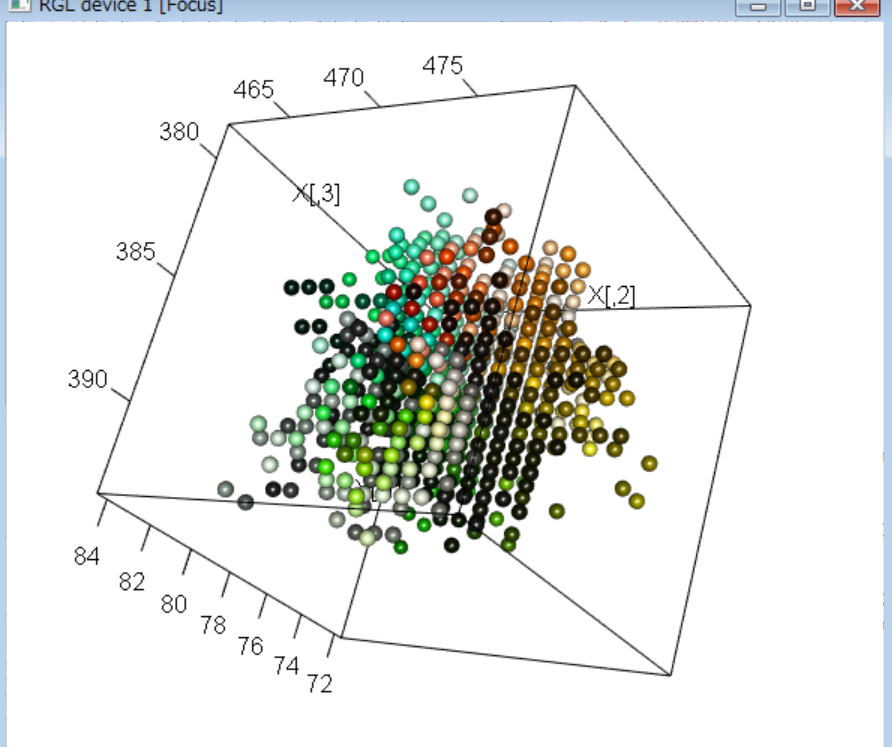
out <- my.cell.walk.dt.multi(n.cell=n.cell,nt=nt,n.iter=n.iter,n.step=n.step,xyz=c(500,500,100))
X <- out[,1:3]
X.st <- t(t(X) - apply(X,2,mean))
X.st <- X.st/max(sqrt(apply(X.st^2,1,sum)))
col <- my.angle.complex.color(my.3d.angles(X.st))
library(rgl)
plot3d(X)
spheres3d(X,radius=0.3,color=col)
