- さて、うんちくは前の記事に任せて、使ってみることにしよう
- まずは、密行列と疎行列のうちの、密行列の扱い
- 密行列
- 形に応じて、7種類の密行列オブジェクトを用意している
- dgeMatrix:普通の実数行列を普通に保管
- dsyMatrix:実対称行列を圧縮しないで保管
- dspMatrix:実対称行列を圧縮保管(三角部分の保管)
- dtrMatrix:実三角行列の非圧縮保管
- dtpMatrix:実三角行列の圧縮保管(三角部分のみ)
- dpoMatrix:Positive semi-definite symmetric realを非圧縮保管
- dppMatrix:Positive semi-definite symmetric realを圧縮保管
- この形式を、計算負荷が小さくなるように、適宜強制変換しながら演算(行列の積、cross積、行列norm、reciprocal condition number(数値計算精度を保つための指標)、LU factorization,これスキー分解、逆行列)を実施するように作ってある
- 分解計算などでは、分解成分を使い捨てにせず、再利用することを前提に保管しながら計算を進める
- 疎行列
- 疎行列保管の基礎
- 非ゼロのセルを、行・列・値の3つ組で保管
- 要素の並べ方は、行でのつながりを優先するか(csr:compressed sparse row)、列でのそれを優先するか(csc:compressed sparse column)、で二通り定められる
- RとMatlabは密行列の保管において、column優先システムを採用
- 形に応じて、4種類の疎行列オブジェクトを用意している
- dgTMatrix: 3つ組の原則での保管形式(順序は適当?圧縮していない?)
- dgCMatrix: csc順に並び替えて(csc形式)の疎行列圧縮保管
- dsCMatrix: 実対称疎行列をcsc形式で圧縮保管
- dtCMatrix: 三角実疎行列をcsc形式で圧縮保管
- ddiMatrix: 対角行列の型もある
- 使ってみる
- Matrix()関数に適当に0がちなデータを与えると、dgeMatrix形式(密行列の形式)としたり、dgCMatrix(疎行列の形式としたりする)
for(i in 1:6){
print(Matrix(sample(0:1,28,replace=TRUE,prob=c(0.5,0.5)),4,7))
}
4 x 7 Matrix of class "dgeMatrix"
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
[1,] 1 1 1 1 0 1 0
[2,] 1 0 1 1 1 1 1
[3,] 1 1 0 1 0 0 0
[4,] 0 1 1 1 1 0 0
4 x 7 sparse Matrix of class "dgCMatrix"
[1,] 1 1 . . 1 1 1
[2,] 1 1 1 . 1 . .
[3,] . . . 1 1 1 .
[4,] . . . . 1 . .
4 x 7 sparse Matrix of class "dgCMatrix"
[1,] . 1 1 1 1 1 .
[2,] . . 1 1 1 1 .
[3,] . 1 . 1 1 . .
[4,] . . . 1 . . .
4 x 7 Matrix of class "dgeMatrix"
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
[1,] 0 0 1 1 1 0 0
[2,] 1 0 0 1 1 1 1
[3,] 0 1 0 0 1 0 1
[4,] 1 1 0 0 0 1 0
4 x 7 Matrix of class "dgeMatrix"
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
[1,] 1 0 0 1 1 0 1
[2,] 1 0 0 1 0 1 0
[3,] 0 1 0 0 0 1 1
[4,] 0 1 1 1 1 0 0
4 x 7 Matrix of class "dgeMatrix"
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
[1,] 1 1 1 0 1 1 0
[2,] 1 0 0 0 0 1 0
[3,] 0 0 1 0 1 1 1
[4,] 0 1 0 1 1 1 1
- 対称行列なら、dsyMatrix,dsCMatrix形式になる
for(i in 1:6){
m <- matrix(sample(0:1,25,replace=TRUE,prob=c(0.8,0.2)),5,5)
m <- m+t(m)
print(Matrix(c(m),5,5))
}
5 x 5 sparse Matrix of class "dsCMatrix"
[1,] . . . 1 1
[2,] . . 1 . .
[3,] . 1 . . .
[4,] 1 . . . .
[5,] 1 . . . 2
5 x 5 sparse Matrix of class "dsCMatrix"
[1,] . . . . .
[2,] . . 1 . 1
[3,] . 1 . 1 .
[4,] . . 1 . 1
[5,] . 1 . 1 .
5 x 5 Matrix of class "dsyMatrix"
[,1] [,2] [,3] [,4] [,5]
[1,] 0 0 1 1 1
[2,] 0 0 0 0 1
[3,] 1 0 2 1 1
[4,] 1 0 1 2 0
[5,] 1 1 1 0 0
5 x 5 sparse Matrix of class "dsCMatrix"
[1,] . 1 2 . .
[2,] 1 . . . 1
[3,] 2 . . . .
[4,] . . . 2 1
[5,] . 1 . 1 2
5 x 5 sparse Matrix of class "dsCMatrix"
[1,] 2 . 1 . .
[2,] . 2 1 . .
[3,] 1 1 . . .
[4,] . . . 2 1
[5,] . . . 1 .
5 x 5 Matrix of class "dsyMatrix"
[,1] [,2] [,3] [,4] [,5]
[1,] 0 1 1 0 0
[2,] 1 2 1 1 1
[3,] 1 1 0 0 0
[4,] 0 1 0 0 1
[5,] 0 1 0 1 0
- 同様に三角行列ならdtrMatrix,dtCMatirx形式になる
for(i in 1:6){
m <- matrix(sample(0:1,25,replace=TRUE,prob=c(0.2,0.8)),5,5)
m[lower.tri(m)] <- 0
print(Matrix(c(m),5,5))
}
5 x 5 sparse Matrix of class "dtCMatrix"
[1,] 1 . 1 1 1
[2,] . . . 1 1
[3,] . . 1 . 1
[4,] . . . 1 1
[5,] . . . . 1
5 x 5 Matrix of class "dtrMatrix"
[,1] [,2] [,3] [,4] [,5]
[1,] 1 1 0 1 1
[2,] . 1 1 1 1
[3,] . . 1 1 1
[4,] . . . 1 1
[5,] . . . . 1
5 x 5 Matrix of class "dtrMatrix"
[,1] [,2] [,3] [,4] [,5]
[1,] 1 1 1 1 1
[2,] . 1 1 0 1
[3,] . . 1 1 1
[4,] . . . 1 0
[5,] . . . . 1
5 x 5 sparse Matrix of class "dtCMatrix"
[1,] 1 1 1 1 .
[2,] . 1 1 . 1
[3,] . . 1 1 1
[4,] . . . 1 .
[5,] . . . . 1
5 x 5 Matrix of class "dtrMatrix"
[,1] [,2] [,3] [,4] [,5]
[1,] 1 1 1 1 1
[2,] . 1 1 1 0
[3,] . . 0 1 1
[4,] . . . 1 1
[5,] . . . . 1
5 x 5 sparse Matrix of class "dtCMatrix"
[1,] 1 1 . . 1
[2,] . . 1 . .
[3,] . . 1 1 1
[4,] . . . 1 1
[5,] . . . . 1
- 実際、igraphパッケージの隣接行列はdgCMatrix形式も使って返ってくるようだ
> library(igraph)
> n.e <- 20
> n.v <- 10
> el <- cbind(sample(1:n.v,n.e,replace=TRUE),sample(1:n.v,n.e,replace=TRUE))
> g <- graph.edgelist(el)
> m <- get.adjacency(g)
> m
10 x 10 sparse Matrix of class "dgCMatrix"
[1,] 1 . 1 . 2 . . . . 1
[2,] . . 1 . . 1 . . . .
[3,] 1 . . . . 1 1 . . .
[4,] . . . . . 2 . . . .
[5,] . . 1 . . . . . . .
[6,] . . . . . 1 . 1 . .
[7,] . . . . . . . . . .
[8,] . 1 . . 1 . . . . .
[9,] . . 1 . . . . . . .
[10,] 2 . . . . . . . . .
> n.e <- 1000
> n.v <- 10
> el <- cbind(sample(1:n.v,n.e,replace=TRUE),sample(1:n.v,n.e,replace=TRUE))
> g <- graph.edgelist(el)
> m <- get.adjacency(g)
> m
10 x 10 sparse Matrix of class "dgCMatrix"
[1,] 6 11 11 4 13 12 15 7 9 8
[2,] 11 8 13 11 6 6 6 14 11 16
[3,] 11 8 14 6 9 7 16 13 17 11
[4,] 8 14 9 14 11 12 14 14 15 10
[5,] 14 7 10 5 7 7 11 10 14 9
[6,] 10 9 5 10 6 9 13 11 5 5
[7,] 7 7 13 8 9 7 9 12 4 11
[8,] 10 4 11 8 8 9 10 8 12 7
[9,] 11 7 14 8 11 13 11 12 14 7
[10,] 10 10 16 10 13 7 9 12 10 13
library(igraph)
n.e <- 20
n.v <- 10
el <- cbind(sample(1:n.v,n.e,replace=TRUE),sample(1:n.v,n.e,replace=TRUE))
g <- graph.edgelist(el)
m <- get.adjacency(g)
m
m.dens <- as(m,"denseMatrix")
m.dens
m2 <- as(m.dens,"sparseMatrix")
m2
> m
10 x 10 sparse Matrix of class "dgCMatrix"
[1,] 2 . 2 . . . . . . .
[2,] . . . . . . . . . .
[3,] . . 1 1 . . . . . .
[4,] . 1 2 1 . 1 . . . .
[5,] . 1 . . 1 . . . 1 .
[6,] . . . . 1 . . . . .
[7,] . . . . . . 1 1 . .
[8,] . . . . . . . 1 . .
[9,] . 1 . . . . . . . .
[10,] . 1 . . . . . . . .
> m.dens <- as(m,"denseMatrix")
> m.dens
10 x 10 Matrix of class "dgeMatrix"
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
[1,] 2 0 2 0 0 0 0 0 0 0
[2,] 0 0 0 0 0 0 0 0 0 0
[3,] 0 0 1 1 0 0 0 0 0 0
[4,] 0 1 2 1 0 1 0 0 0 0
[5,] 0 1 0 0 1 0 0 0 1 0
[6,] 0 0 0 0 1 0 0 0 0 0
[7,] 0 0 0 0 0 0 1 1 0 0
[8,] 0 0 0 0 0 0 0 1 0 0
[9,] 0 1 0 0 0 0 0 0 0 0
[10,] 0 1 0 0 0 0 0 0 0 0
> m2 <- as(m.dens,"sparseMatrix")
> m2
10 x 10 sparse Matrix of class "dgCMatrix"
[1,] 2 . 2 . . . . . . .
[2,] . . . . . . . . . .
[3,] . . 1 1 . . . . . .
[4,] . 1 2 1 . 1 . . . .
[5,] . 1 . . 1 . . . 1 .
[6,] . . . . 1 . . . . .
[7,] . . . . . . 1 1 . .
[8,] . . . . . . . 1 . .
[9,] . 1 . . . . . . . .
[10,] . 1 . . . . . . . .
- 疎行列形式の値の格納方法はどうなっているのか?
- 以下に示すように、m2@iには、各列について、第何行に0以外の値が入っているのかが格納されている(0始まり。第0列は第0行、第1列は第3,4,8,9列…。またm2@pには、0以外の値が通しでベクトル化してあるのだが、そのベクトルの番地のうち、各列の最初の値に相当するのはいくつか、が格納されている。最初の0は、ベクトルの第0番地が、第0列の最初の値、と宣言してあり、次の1はベクトルの第1番地が、第1列の最初の値と宣言してある、最初の値が0で次の値が1だから、第1列には1個しか非0の値が入っていないこともそこからわかる。第2列には4個の非0があるので、@pの第3番の値は、1+4=5
> str(m2)
Formal class 'dgCMatrix' [package "Matrix"] with 6 slots
..@ i : int [1:17] 0 3 4 8 9 0 2 3 2 3 ...
..@ p : int [1:11] 0 1 5 8 10 12 13 14 16 17 ...
..@ Dim : int [1:2] 10 10
..@ Dimnames:List of 2
.. ..$ : NULL
.. ..$ : NULL
..@ x : num [1:17] 2 1 1 1 1 2 1 2 1 1 ...
..@ factors : list()
> m2
10 x 10 sparse Matrix of class "dgCMatrix"
[1,] 2 . 2 . . . . . . .
[2,] . . . . . . . . . .
[3,] . . 1 1 . . . . . .
[4,] . 1 2 1 . 1 . . . .
[5,] . 1 . . 1 . . . 1 .
[6,] . . . . 1 . . . . .
[7,] . . . . . . 1 1 . .
[8,] . . . . . . . 1 . .
[9,] . 1 . . . . . . . .
[10,] . 1 . . . . . . . .
> m2@i
[1] 0 3 4 8 9 0 2 3 2 3 4 5 3 6 6 7 4
> m2@p
[1] 0 1 5 8 10 12 13 14 16 17 17
- 3つ組表示。疎行列は行番号、列番号、値の3つ組が基本だが、その形式で表示するには
> summary(m2)
10 x 10 sparse Matrix of class "dgCMatrix", with 17 entries
i j x
1 1 1 2
2 4 2 1
3 5 2 1
4 9 2 1
5 10 2 1
6 1 3 2
7 3 3 1
8 4 3 2
9 3 4 1
10 4 4 1
11 5 5 1
12 6 5 1
13 4 6 1
14 7 7 1
15 7 8 1
16 8 8 1
17 5 9 1
- 疎行列ならばimage()関数でプロットするのも軽い
- さすがに、以下の例(1辺の長さが1の16次元超立方格子グラフの隣接行列を疎行列で表したもののimage()プロット)
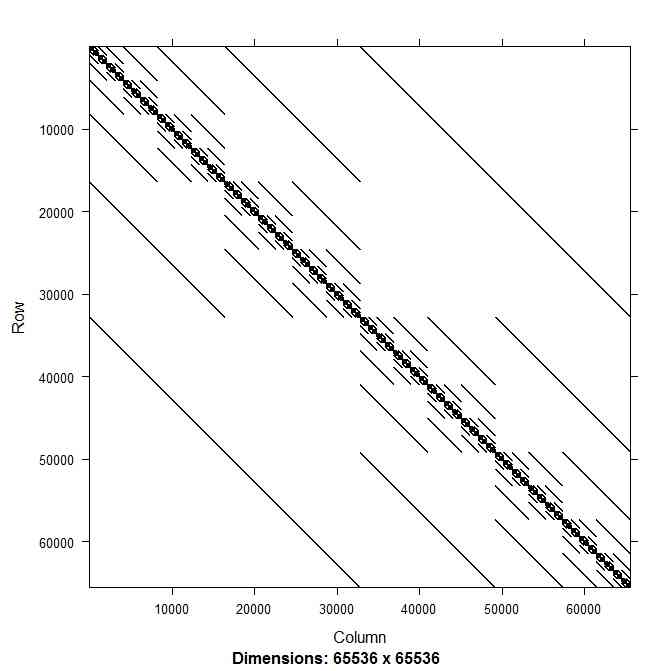
library(Matrix)
library(igraph)
g.lattice <- graph.lattice(length=2, dim=16)
m.lattice <- get.adjacency(g.lattice)
image(m.lattice)
- この規則的な多次元単位格子グラフの隣接行列は、1次元の場合をスタートして、それを疎行列として、単位行列と列バインド・行バインドすることを再帰的に行うことによって作ることができる
- k次元を一つ上げるときには、k次元格子2つを上下に重ねて、対応する点ペア間にエッジを張ればよい。k次元格子の行列を対角に置くのが、2つのk次元格子を持ってくることに相当し、残りのスペースに単位行列を置くのが、2つのk次元格子の対応点間エッジを張ることに相当する
- cBind(),rBind()という疎行列用の連結関数を使っていることに注意
library(Matrix)
library(igraph)
dfs <- 1:8
Lattice.seq.jimichi <- list()
for(i in 1:length(dfs)){
g.lattice <- graph.lattice(length=2, dim=dfs[i])
Lattice.seq.jimichi[[i]] <- get.adjacency(g.lattice)
}
g.lattice <- graph.lattice(length=2, dim=8)
m.lattice <- get.adjacency(g.lattice)
image(m.lattice)
my.serial.lattice2 <- function(X){
tmp <- cBind(X,Matrix(diag(rep(1,nrow(X)))))
rBind(tmp, cBind(Matrix(diag(rep(1,nrow(X)))),X))
}
g.lattice <- graph.lattice(length=2, dim=1)
m.lattice1 <- get.adjacency(g.lattice)
Lattice.seq <- list()
Lattice.seq[[1]] <- m.lattice1
for(i in 2:length(dfs)){
Lattice.seq[[i]] <- my.serial.lattice2(Lattice.seq[[i-1]])
}
- さらに言えば、入れ子になっているので、各次元の行列をいちいち作る必要はなくて、最大次元の行列を作り、その部分を取り出せばよいことになる
library(Matrix)
library(igraph)
dfs <- 1:8
Lattice.seq.jimichi <- list()
for(i in 1:length(dfs)){
g.lattice <- graph.lattice(length=2, dim=dfs[i])
Lattice.seq.jimichi[[i]] <- get.adjacency(g.lattice)
}
g.lattice <- graph.lattice(length=2, dim=8)
m.lattice <- get.adjacency(g.lattice)
image(m.lattice)
my.serial.lattice2 <- function(X){
tmp <- cBind(X,Matrix(diag(rep(1,nrow(X)))))
rBind(tmp, cBind(Matrix(diag(rep(1,nrow(X)))),X))
}
g.lattice <- graph.lattice(length=2, dim=1)
m.lattice1 <- get.adjacency(g.lattice)
Lattice.seq <- list()
Lattice.seq[[1]] <- m.lattice1
dfs <- 1:8
for(i in 2:length(dfs)){
Lattice.seq[[i]] <- my.serial.lattice2(Lattice.seq[[i-1]])
}
Lattice.seq. <- m.lattice1
for(i in 1:10){
Lattice.seq. <- my.serial.lattice2(Lattice.seq.)
}
Lattice.seq.3 <- list()
for(i in 1:8){
Lattice.seq.3[[i]] <- Lattice.seq.[1:2^i,1:2^i]
}
for(i in 1:8){
print(identical(Lattice.seq[[i]],Lattice.seq.3[[i]]))
}
