- 昨日、移動のことをメモした
- 結晶というのは、"Mathematical model for ideal crystal (or perfect crystal) in R^d is a set Λ, which is formed by a finite number of shifted copies of a single lattice L. Formally, Λ is a perfect crystal if Λ = L + S, where S is a finite set of translations. Since lattices satisfy L − L ⊂ L and a perfect crystal satisfies Λ − Λ ⊂ Λ − S, they are both Meyer sets. The Meyer concept of quasicrystals thus generalizes the classical definition of crystals. Perfect crystal is however a periodic set, i.e. Λ + x ⊂ Λ for any x ∈ L, and therefore it is not a suitable model for quasicrystalline materials, which reveal rotational symmetries ncompatible with periodicity. We shall now describe a large class of Meyer sets which are not invariant under translation."とこちらにあるように、「シフト」したときに重ね合わせられる
- このことと「移動」とその距離の評価という点から考えると、「シフトして重ねあわせた」とき、その移動量は、すべての点が同じだけ動いたものとして、その量を点の数で倍するとみなしてもよいし。重なった点は不動で、端の点だけが長距離移動したと考えてもよい
- どちらの計算方法でも、移動距離の総量は変わらない、という評価法である
- 点集合の移動量の総量も同様に考えることができる
- では、各点の移動量の総量よりも移動のベクトルの具合について興味があるときはどうなるだろうか
- 総量だけで比べれば同じだけれど、平行移動と端点のみの長距離移動は区別したい
- そのようにするには、すべての点の移動の総和や総平均に着目するのではなくて、その分散・ばらつきに注意するのがよい
- そんなことを考えて作ったのが昨日の記事の、「内積」利用の評価指標
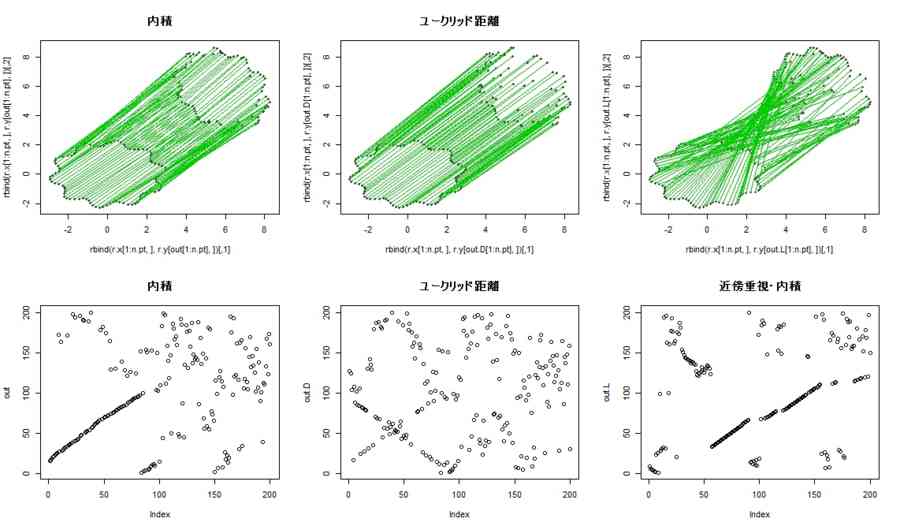
- すべてのベクトルが似たような方向・長さになっているかどうかを強調したのが次の図であり、そのベクトルを散布図表示もしてみた
library(geometry)
d <- 2
n.pt <- 100
t <- seq(from=0,to=1,length=n.pt)*2*pi
R.X <- 0.2*sin(t*10)+2.1
X <- cbind(R.X*cos(t)*1.3,R.X*sin(t))
Y <- cbind(X[,1],X[,2])
theta <- pi/3
Rot <- matrix(c(cos(theta),sin(theta),-sin(theta),cos(theta)),2,2)
Y <- Y %*% Rot
Y <- (Y-min(Y)) + 3
delaunay.X <- delaunayn(X)
plot(X)
delaunay.X.2 <- rbind(delaunay.X[,1:2],delaunay.X[,2:3],delaunay.X[,c(3,1)])
segments(X[delaunay.X.2[,1],1],X[delaunay.X.2[,1],2],X[delaunay.X.2[,2],1],X[delaunay.X.2[,2],2])
segments(X[delaunay.X.2[,1],1],X[delaunay.X.2[,1],2],X[delaunay.X.2[,2],1],X[delaunay.X.2[,2],2])
X.hull <- t(convhulln(X))
Y.hull <- t(convhulln(Y))
plot(X,pch=20)
segments(X[X.hull[1,],1],X[X.hull[1,],2],X[X.hull[2,],1],X[X.hull[2,],2])
plot(Y,pch=20)
segments(Y[Y.hull[1,],1],Y[Y.hull[1,],2],Y[Y.hull[2,],1],Y[Y.hull[2,],2])
X.h <- X[unique(c(X.hull)),]
Y.h <- Y[unique(c(Y.hull)),]
plot(X.h)
delaunay.X.h <- delaunayn(X.h)
plot(X.h)
delaunay.X.h.2 <- rbind(delaunay.X.h[,1:2],delaunay.X.h[,2:3],delaunay.X.h[,c(3,1)])
segments(X.h[delaunay.X.h.2[,1],1],X.h[delaunay.X.h.2[,1],2],X.h[delaunay.X.h.2[,2],1],X.h[delaunay.X.h.2[,2],2])
plot(Y.h)
delaunay.Y.h <- delaunayn(Y.h)
plot(Y.h)
delaunay.Y.h.2 <- rbind(delaunay.Y.h[,1:2],delaunay.Y.h[,2:3],delaunay.Y.h[,c(3,1)])
segments(Y.h[delaunay.Y.h.2[,1],1],Y.h[delaunay.Y.h.2[,1],2],Y.h[delaunay.Y.h.2[,2],1],Y.h[delaunay.Y.h.2[,2],2])
my.simplex.volume <- function(V,factorial=FALSE){
n <- length(V[1,])
K <- matrix(V[1:n,],ncol=n)
K. <- t(K)-V[n+1,]
if(factorial){
return(det(K.)/factorial(n))
}else{
return(det(K.))
}
}
my.delaunay.vol <- function(a,X){
tmp <- matrix(X[a,],ncol=length(X[1,]))
my.simplex.volume(tmp)
}
spx.vol.x <- apply(delaunay.X.h,1,my.delaunay.vol,X.h)
spx.vol.y <- apply(delaunay.Y.h,1,my.delaunay.vol,Y.h)
spx.vol.x.st <- abs(spx.vol.x)/sum(abs(spx.vol.x))
spx.vol.y.st <- abs(spx.vol.y)/sum(abs(spx.vol.y))
n.r.pt <- 100
r.x <- r.y <- matrix(0,n.r.pt,d)
spx.vol.x.st.cum <- cumsum(spx.vol.x.st)
x.rd <- round(spx.vol.x.st.cum*n.r.pt)
r.n.x <- c(x.rd[1],diff(x.rd))
r.n.x
spx.vol.y.st.cum <- cumsum(spx.vol.y.st)
y.rd <- round(spx.vol.y.st.cum*n.r.pt)
r.n.y <- c(y.rd[1],diff(y.rd))
r.n.y
tri.id.x <- sample(1:length(spx.vol.x.st),n.r.pt,replace=TRUE,prob=spx.vol.x.st)
tri.id.y <- sample(1:length(spx.vol.y.st),n.r.pt,replace=TRUE,prob=spx.vol.y.st)
library(MCMCpack)
cnt <- 1
for(i in 1:length(spx.vol.x.st)){
tmp.id <- cnt:(cnt+r.n.x[i]-1)
cnt <- cnt+r.n.x[i]
tmp.n <- r.n.x[i]
if(tmp.n>0){
tmp.r <- rdirichlet(tmp.n,rep(1,length(delaunay.X.h[i,])))
tmp.X <- tmp.r %*% X.h[delaunay.X.h[i,],]
r.x[tmp.id,] <- tmp.X
}
}
cnt <- 1
for(i in 1:length(spx.vol.y.st)){
tmp.id <- cnt:(cnt+r.n.y[i]-1)
cnt <- cnt+r.n.y[i]
tmp.n <- r.n.y[i]
if(tmp.n>0){
tmp.r <- rdirichlet(tmp.n,rep(1,length(delaunay.Y.h[i,])))
tmp.Y <- tmp.r %*% Y.h[delaunay.Y.h[i,],]
r.y[tmp.id,] <- tmp.Y
}
}
plot(X.h)
delaunay.X.h.2 <- rbind(delaunay.X.h[,1:2],delaunay.X.h[,2:3],delaunay.X.h[,c(3,1)])
segments(X.h[delaunay.X.h.2[,1],1],X.h[delaunay.X.h.2[,1],2],X.h[delaunay.X.h.2[,2],1],X.h[delaunay.X.h.2[,2],2])
points(r.x,pch=20,col=2)
plot(Y.h)
delaunay.Y.h.2 <- rbind(delaunay.Y.h[,1:2],delaunay.Y.h[,2:3],delaunay.Y.h[,c(3,1)])
segments(Y.h[delaunay.Y.h.2[,1],1],Y.h[delaunay.Y.h.2[,1],2],Y.h[delaunay.Y.h.2[,2],1],Y.h[delaunay.Y.h.2[,2],2])
points(r.y,pch=20,col=2)
r.x <- rbind(X,r.x)
r.y <- rbind(Y,r.y)
out <- my.move(r.x,r.y)
out.D <- my.move.D(r.x,r.y)
out.L <- my.move.L(r.x,r.y)
par(mfcol=c(2,3))
plot(rbind(r.x[1:n.pt,],r.y[out[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])),main="内積")
for(i in 1:n.pt){
segments(r.x[i,1],r.x[i,2],r.y[out[i],1],r.y[out[i],2],col=3)
}
plot(out,main="内積")
plot(rbind(r.x[1:n.pt,],r.y[out.D[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])),main="ユークリッド距離")
for(i in 1:n.pt){
segments(r.x[i,1],r.x[i,2],r.y[out.D[i],1],r.y[out.D[i],2],col=3)
}
plot(out.D,main="ユークリッド距離")
plot(rbind(r.x[1:n.pt,],r.y[out.L[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])))
for(i in 1:n.pt){
segments(r.x[i,1],r.x[i,2],r.y[out.L[i],1],r.y[out.L[i],2],col=3,main="近傍重視・内積")
}
plot(out.L,main="近傍重視・内積")
par(mfcol=c(1,1))
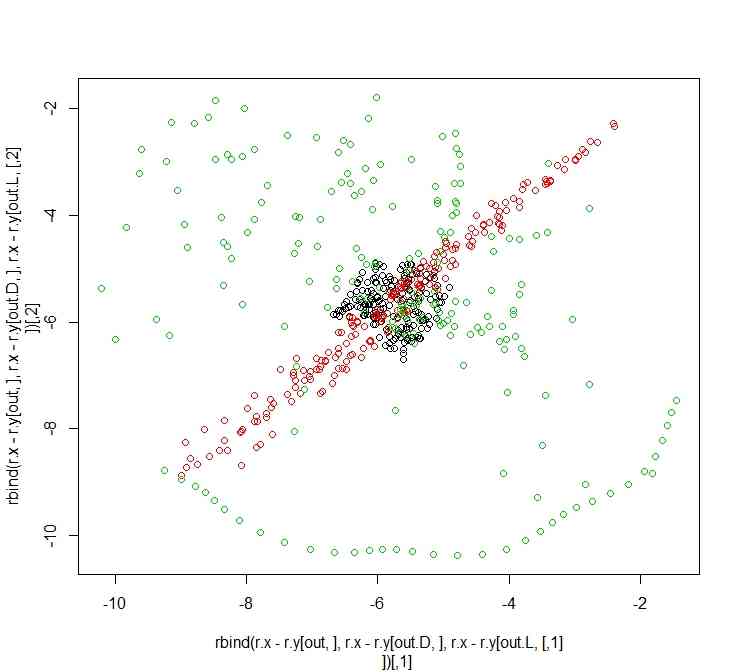
plot(rbind(r.x-r.y[out,],r.x-r.y[out.D,],r.x-r.y[out.L,]),col=rep(1:3,each=length(r.x[,1])))
plot(rbind((r.x-r.y[out,])[1:n.pt,],(r.x-r.y[out.D,])[1:n.pt,],(r.x-r.y[out.L,])[1:n.pt,]),col=rep(1:3,each=n.pt))
out.out <- my.move.D(r.y[out[1:n.pt],],r.y[1:n.pt,])
out.out.D <- my.move.D(r.y[out.D[1:n.pt],],r.y[1:n.pt,])
out.out.L <- my.move.D(r.y[out.L[1:n.pt],],r.y[1:n.pt,])
par(mfcol=c(1,3))
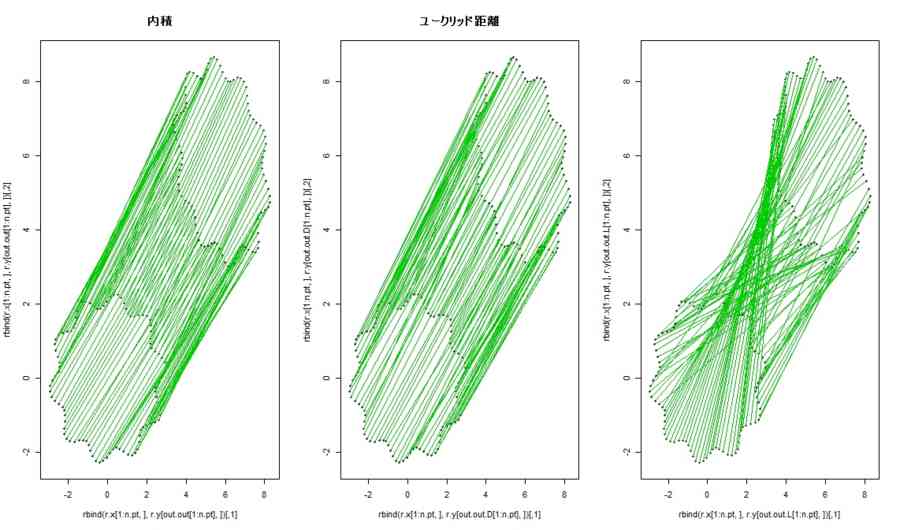
plot(rbind(r.x[1:n.pt,],r.y[out.out[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])),main="内積")
for(i in 1:n.pt){
segments(r.x[i,1],r.x[i,2],r.y[out.out[i],1],r.y[out.out[i],2],col=3)
}
plot(rbind(r.x[1:n.pt,],r.y[out.out.D[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])),main="ユークリッド距離")
for(i in 1:n.pt){
segments(r.x[i,1],r.x[i,2],r.y[out.out.D[i],1],r.y[out.out.D[i],2],col=3)
}
plot(rbind(r.x[1:n.pt,],r.y[out.out.L[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])))
for(i in 1:n.pt){
segments(r.x[i,1],r.x[i,2],r.y[out.out.L[i],1],r.y[out.out.L[i],2],col=3,main="近傍重視・内積")
}
par(mfcol=c(1,1))
par(mfcol=c(1,2))
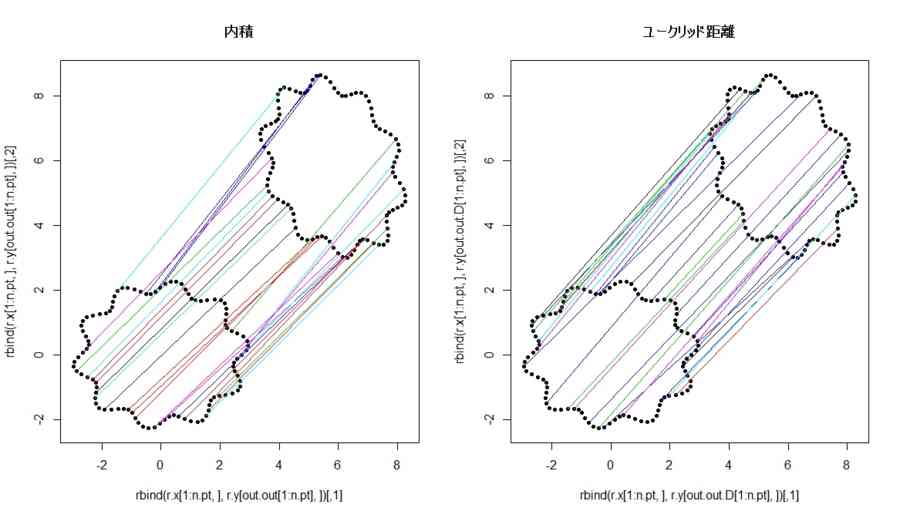
plot(rbind(r.x[1:n.pt,],r.y[out.out[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])),main="内積")
for(i in 1:n.pt){
if(runif(1) < 0.3){
segments(r.x[i,1],r.x[i,2],r.y[out.out[i],1],r.y[out.out[i],2],col=sample(1:6,1))
}
}
plot(rbind(r.x[1:n.pt,],r.y[out.out.D[1:n.pt],]),pch=20,col=rep(1:2,each=length(r.x[,1])),main="ユークリッド距離")
for(i in 1:n.pt){
if(runif(1)<0.3){
segments(r.x[i,1],r.x[i,2],r.y[out.out.D[i],1],r.y[out.out.D[i],2],col=sample(1:6,1))
}
}
par(mfcol=c(1,1))
my.move <- function(x,y,w.x=NULL,w.y=NULL){
if(!is.matrix(x)){
x <- matrix(x,ncol=1)
}
if(!is.matrix(y)){
y < matrix(y,ncol=1)
}
if(is.null(w.x)){
w.x <- rep(1,length(x[,1]))
}
if(is.null(w.y)){
w.y <- rep(1,length(y[,1]))
}
D <- x %*% t(y)
D <- D-min(D)
xy <- rbind(x,y)
m.xy <- apply(xy,2,mean)
x.d <- t(t(x)-m.xy)
y.d <- t(t(y)-m.xy)
W <- outer(w.x,w.y,"*")
D <- D * W
as.vector(solve_LSAP(D,maximum=TRUE))
}
my.move.D <- function(x,y,w.x=NULL,w.y=NULL){
if(!is.matrix(x)){
x <- matrix(x,ncol=1)
}
if(!is.matrix(y)){
y < matrix(y,ncol=1)
}
if(is.null(w.x)){
w.x <- rep(1,length(x[,1]))
}
if(is.null(w.y)){
w.y <- rep(1,length(y[,1]))
}
D <- (as.matrix(dist(rbind(x,y))))[1:length(x[,1]),(1+length(x[,1])):(2*length(x[,1]))]
xy <- rbind(x,y)
m.xy <- apply(xy,2,mean)
W <- outer(w.x,w.y,"*")
D <- D * W
as.vector(solve_LSAP(D,maximum=FALSE))
}
my.move.L <- function(x,y,w.x=NULL,w.y=NULL){
if(!is.matrix(x)){
x <- matrix(x,ncol=1)
}
if(!is.matrix(y)){
y < matrix(y,ncol=1)
}
if(is.null(w.x)){
w.x <- rep(1,length(x[,1]))
}
if(is.null(w.y)){
w.y <- rep(1,length(y[,1]))
}
D <- x %*% t(y)
D <- D-min(D)
xy <- rbind(x,y)
m.xy <- apply(xy,2,mean)
x.d <- t(t(x)-m.xy)
y.d <- t(t(y)-m.xy)
L.x <- sqrt(apply(x.d^2,1,sum))
L.y <- sqrt(apply(y.d^2,1,sum))
LL <- outer(L.x,L.x,"*")
L.q <- quantile(LL,0.05)
LL.q <- sign(LL < L.q)
W <- outer(w.x,w.y,"*")
D <- D * W * LL.q
as.vector(solve_LSAP(D,maximum=TRUE))
}



