- 2. 曲面論
- パラメタ表示された曲面を描いてみよう
- 曲面とは
 の部分集合で、局所的に2つのパラメタで表されるもの
の部分集合で、局所的に2つのパラメタで表されるもの
- 2つのパラメタの偏微分が1次独立であると「都合がよい」
- 閉曲面は有界(含まれている範囲が有限)で、境界がない曲面
- 裏と表。曲面が空間内である部分を囲んでいると、その曲面には囲んでいる空間側とそうでない側の区別(裏表)ができるが、メビウスの輪にはそれがない→単側曲面、向き付け不可能曲面
- 向き付け不可能な閉曲面は存在しない
- 自身と交差する曲面は、向き付け不可能な閉曲面とみることもできる→クラインの壺
- 自己交差のところでは、同じ点に、2つまたはそれ以上の枚数の曲面が通過する。それぞれの曲面が「曲面の条件」を満たしていること
- 曲面を2パラメタで表したとき、それぞれのパラメタで偏微分して接ベクトルが2つ得られる。これらが1次独立なとき、この2つの接ベクトルを基底とした線形部分空間が取れて、これが接平面(Rで、2パラメタ表現し、その式をderiv()関数で微分して、この様子を見てみる
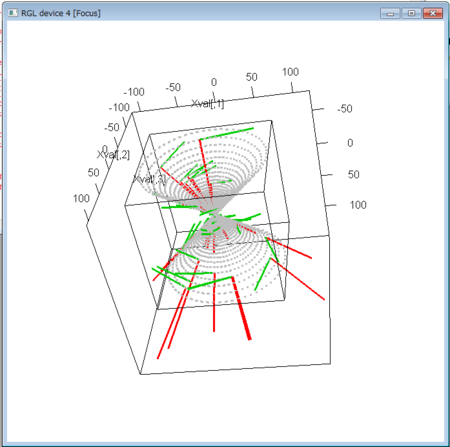
- さらに、法線ベクトルは、2つの接ベクトルの外積ベクトルであるから、外積ベクトルの計算は、こちらの記事で書いたRの関数を使って、付加してみる

x<-y<-seq(from=-10,to=10,length=20)
xy<-expand.grid(x,y)
a<-runif(1)
b<-runif(1)
c<-runif(1)
d<-runif(1)
z<-(d-a*xy[,1]-b*xy[,2])/c
xyz<-cbind(xy,z)
plot3d(xyz)
u<-v<-seq(from=-1,to=1,length=100)*pi
uv<-expand.grid(u,v)
x<-cos(uv[,1])*cos(uv[,2])
y<-cos(uv[,1])*sin(uv[,2])
z<-sin(uv[,1])
open3d()
plot3d(x,y,z,type="l")
a<-runif(1)
b<-runif(1)
c<-runif(1)
xlim<-ylim<-zlim<-c(-max(a,b,c),max(a,b,c))
open3d()
plot3d(a*x,b*y,c*z,type="l",xlim=xlim,ylim=ylim,zlim=zlim)
u<-v<-seq(from=-1,to=1,length=100)*pi
uv<-expand.grid(u,v)
x<-a*cosh(uv[,1])*cos(uv[,2])
y<-b*cosh(uv[,1])*sin(uv[,2])
z<-c*sinh(uv[,1])
open3d()
xlim<-ylim<-zlim<-range(c(x,y,z))
plot3d(x,y,z,xlim=xlim,ylim=ylim,zlim=zlim)
u<-v<-seq(from=-1,to=1,length=100)*pi
uv<-expand.grid(u,v)
x<-a*cosh(uv[,1])*cos(uv[,2])
y<-b*cosh(uv[,1])*sin(uv[,2])
z<-c*cosh(uv[,1])
z2<--c*z
open3d()
x<-c(x,x)
y<-c(y,y)
z<-c(z,z2)
xlim<-ylim<-zlim<-range(c(x,y,z))
plot3d(x,y,z,xlim=xlim,ylim=ylim,zlim=zlim)
x<-a*uv[,1]
y<-b*uv[,2]
z<-uv[,1]^2+uv[,2]^2
xlim<-ylim<-zlim<-range(c(x,y,z))
open3d()
plot3d(x,y,z,xlim=xlim,ylim=ylim,zlim=zlim)
x<-a*uv[,1]
y<-b*uv[,2]
z<-uv[,1]^2-uv[,2]^2
open3d()
xlim<-ylim<-zlim<-range(c(x,y,z))
plot3d(x,y,z,xlim=xlim,ylim=ylim,zlim=zlim)
R<-runif(1)
r<-runif(1)*R
x<-(R+r*cos(uv[,1]))*cos(uv[,2])
y<-(R+r*cos(uv[,1]))*sin(uv[,2])
z<-r*sin(uv[,1])
xlim<-ylim<-zlim<-range(c(x,y,z))
open3d()
plot3d(x,y,z,xlim=xlim,ylim=ylim,zlim=zlim)
a<-runif(1)
x<-uv[,1]*cos(uv[,2])
y<-uv[,1]*sin(uv[,2])
z<-a*uv[,2]
xlim<-ylim<-zlim<-range(c(x,y,z))
open3d()
plot3d(x,y,z)
paramlist<-c("u1","u2")
X<-list()
X[[1]]<-expression(cosh(u1)*cos(u2))
X[[2]]<-expression(cosh(u1)*sin(u2))
X[[3]]<-expression(sinh(u1))
dX<-list()
for(i in 1:length(paramlist)){
dX[[i]]<-list()
for(j in 1:length(X)){
dX[[i]][[j]]<-deriv(X[[j]],paramlist[i])
}
}
tmpu1<-tmpu2<-seq(from=-5,to=5,length=100)
u12<-expand.grid(tmpu1,tmpu2)
Xval<-matrix(0,length(u12[,1]),length(X))
for(i in 1:length(Xval[,1])){
for(j in 1:length(Xval[1,])){
u1<-u12[i,1]
u2<-u12[i,2]
Xval[i,j]<-eval(X[[j]])
}
}
dXval<-list()
for(i in 1:length(paramlist)){
dXval[[i]]<-matrix(0,length(u12[,1]),length(X))
for(j in 1:length(dXval[[i]][,1])){
for(k in 1:length(dXval[[i]][1,])){
u1<-u12[j,1]
u2<-u12[j,2]
dXval[[i]][j,k]<-attr(eval(dX[[i]][[k]]),"gradient")[1]
}
}
}
plot3d(Xval,col="gray")
NselectedPt<-100
selected<-sample(1:length(Xval[,1]),NselectedPt)
segs1<-segs2<-matrix(0,NselectedPt*2,length(X))
for(i in 1:NselectedPt){
tmpX<-Xval[selected[i],]
tmpdX1<-dXval[[1]][selected[i],]
tmpdX2<-dXval[[2]][selected[i],]
segs1[2*i-1,]<-tmpX
segs1[2*i,]<-tmpX+tmpdX1
segs2[2*i-1,]<-tmpX
segs2[2*i,]<-tmpX+tmpdX2
}
segments3d(segs1,col=2,lwd=3)
segments3d(segs2,col=3,lwd=3)
library(MCMCpack)
detComplex<-function(M){
e.out<-eigen(M)
prod(e.out[[1]])
}
ExteriorProduct<-function(V){
ret<-rep(0,length(V[1,]))
for(i in 1:length(ret)){
ret[i]<-(-1)^(i+1)*detComplex(V[,-i])
}
ret
}
n<-7
X<-matrix(complex(real = rdirichlet(1,rep(1,n^2)), imaginary = rdirichlet(1,rep(1,n^2))),ncol=n)
X<-t(Conj(X))%*%X
detX<-detComplex(X)
detX
s<-sample(1:n,(n-1))
ep<-ExteriorProduct(X[s,])
ep
sum(X[-s,]*ep)
detComplex(X)
sum(X[-s,]*ep)-detComplex(X)
paramlist<-c("u1","u2")
X<-list()
X[[1]]<-expression(cosh(u1)*cos(u2))
X[[2]]<-expression(cosh(u1)*sin(u2))
X[[3]]<-expression(sinh(u1))
dX<-list()
for(i in 1:length(paramlist)){
dX[[i]]<-list()
for(j in 1:length(X)){
dX[[i]][[j]]<-deriv(X[[j]],paramlist[i])
}
}
tmpu1<-tmpu2<-seq(from=-5,to=5,length=100)
u12<-expand.grid(tmpu1,tmpu2)
Xval<-matrix(0,length(u12[,1]),length(X))
for(i in 1:length(Xval[,1])){
for(j in 1:length(Xval[1,])){
u1<-u12[i,1]
u2<-u12[i,2]
Xval[i,j]<-eval(X[[j]])
}
}
dXval<-list()
for(i in 1:length(paramlist)){
dXval[[i]]<-matrix(0,length(u12[,1]),length(X))
for(j in 1:length(dXval[[i]][,1])){
for(k in 1:length(dXval[[i]][1,])){
u1<-u12[j,1]
u2<-u12[j,2]
dXval[[i]][j,k]<-attr(eval(dX[[i]][[k]]),"gradient")[1]
}
}
}
plot3d(Xval,col="gray")
NselectedPt<-100
selected<-sample(1:length(Xval[,1]),NselectedPt)
segs1<-segs2<-segs3<-matrix(0,NselectedPt*2,length(X))
for(i in 1:NselectedPt){
tmpX<-Xval[selected[i],]
tmpdX1<-dXval[[1]][selected[i],]
tmpdX2<-dXval[[2]][selected[i],]
segs1[2*i-1,]<-tmpX
segs1[2*i,]<-tmpX+tmpdX1
segs2[2*i-1,]<-tmpX
segs2[2*i,]<-tmpX+tmpdX2
ep.out<-ExteriorProduct(rbind(tmpdX2,tmpdX1))
segs3[2*i-1,]<-tmpX
segs3[2*i,]<-tmpX+ep.out/sqrt(sum(ep.out^2))*sqrt(sum(tmpdX2^2))
}
segments3d(segs1,col=2,lwd=3)
segments3d(segs2,col=3,lwd=3)
segments3d(segs3,col=4,lwd=3)
の部分集合で、局所的に2つのパラメタで表されるもの


