- 球面上にランダムさが分布しているとはどういうことだろうか、という話
- 第一資料はこちらを使う。『量子重力理論』とのかかわりが考えられるとか、広がりが大きそうな予感
- まず、背景知識など
- 平面グラフ
-
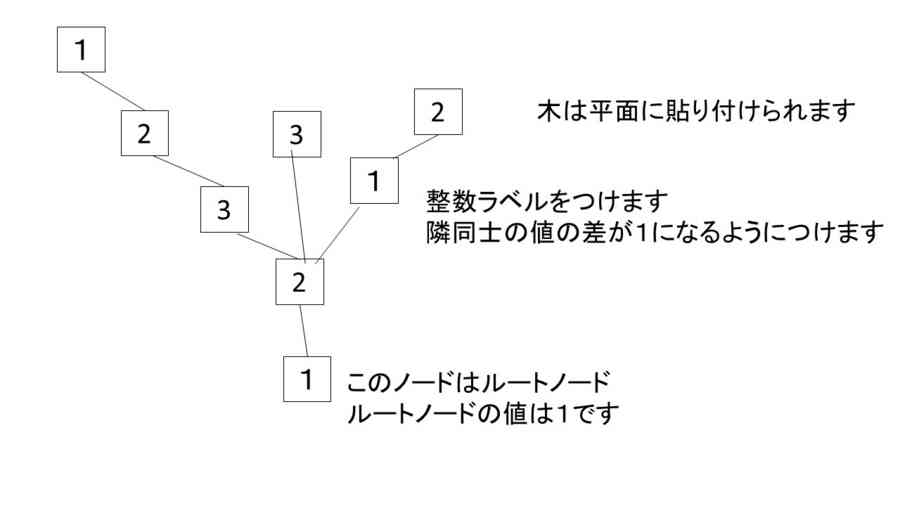
- 三角形分割と木グラフとの対応もある
- この場合は、三角形分割はEulerian triangulationとなっていることが条件。このEulerian triangulationはすべての頂点の次数が偶数であるようなそれ
- この場合は、2色に塗り分けられることが知られている
- 今、2色に塗り分けられた三角形で敷き詰められた平面グラフ(Eulerian triangulation)があったとする。外周の1辺をとり、それに向きを決めることでグラフ全体のエッジに自然に向きを入れることができる
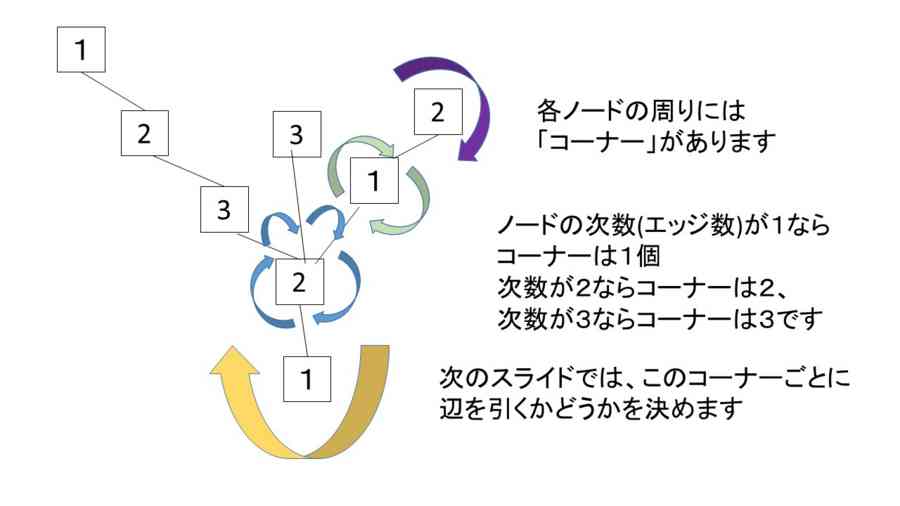
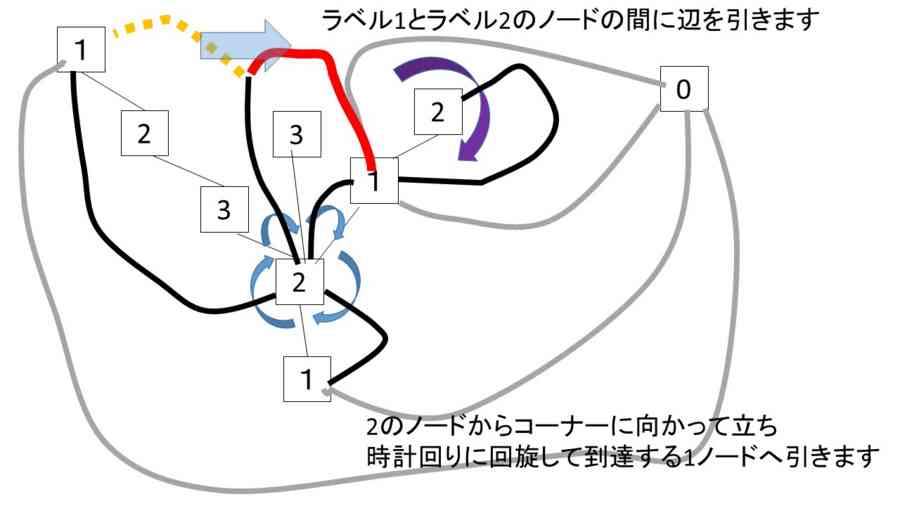
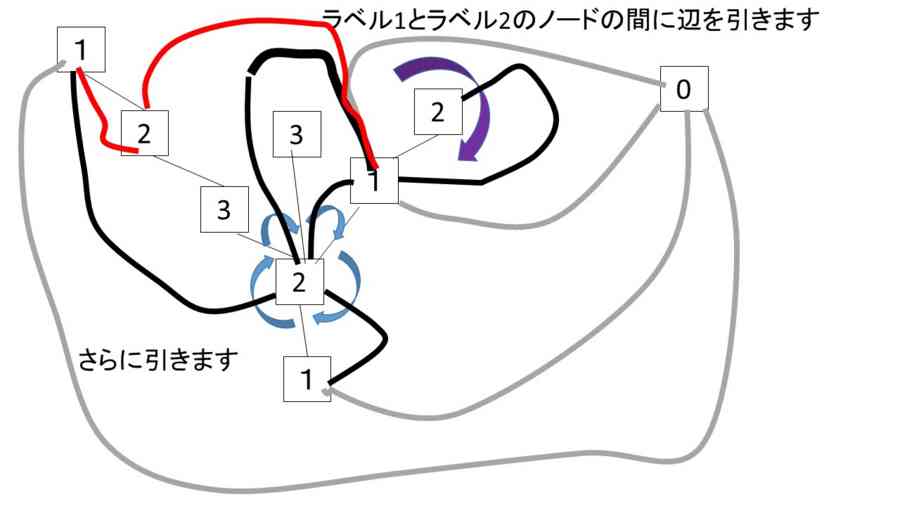
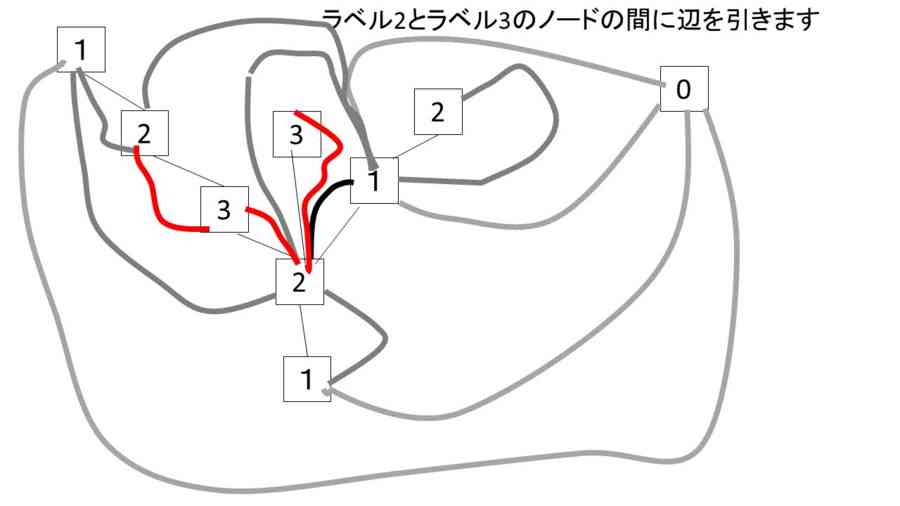
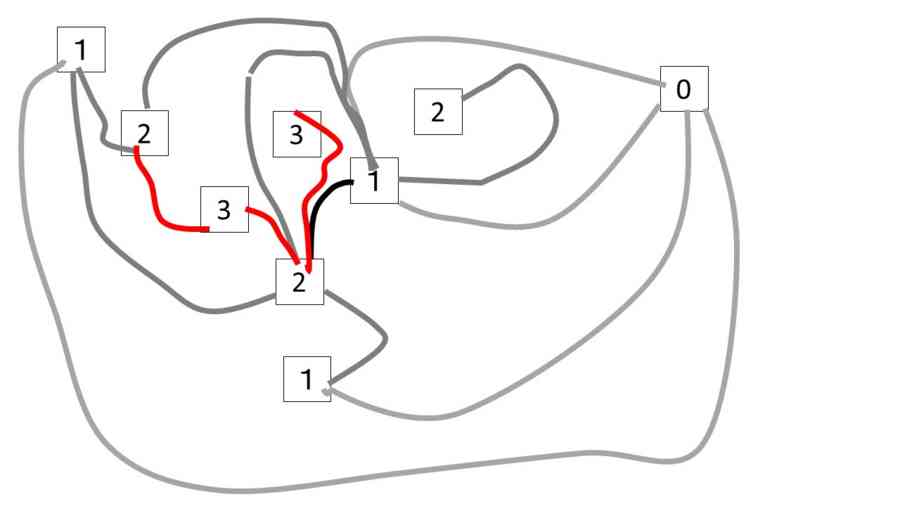
- ここで初めに定めた辺の始点(ルートノード)からの距離をラベルとしてノードに与えると、すべての三角形にn,n+1,n+2というラベルがつく。ルートノードを含む三角形と同じ色の三角形について、n+1->n+2のエッジを取り出すと、それらは、木を成す。そして、そのノードには整数のラベルがついていて、隣り合うノードのラベルの値の差は必ず1である。これをEuler triangulationのWell-labeled treeという(ルートノードは取り去っておく)
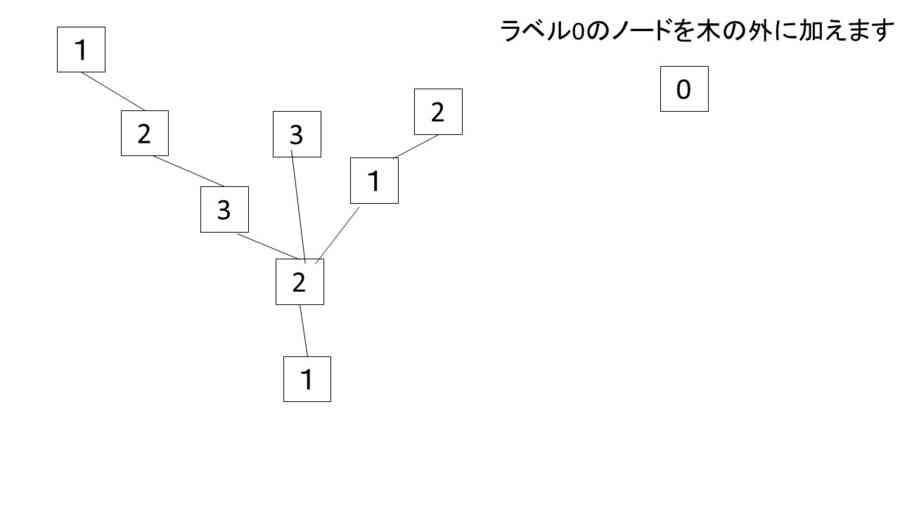
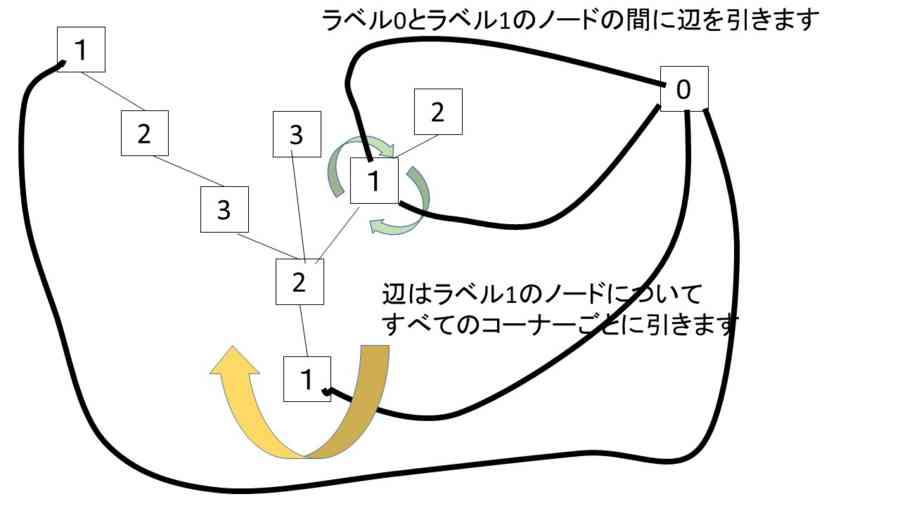
- 今、Well-labeled treeがあったときに、すべてのエッジは、同色の三角形のn+1 -> n+2 の辺に相当しているので、nに相当するノードをその木から探して結べば、Well-labeled treeからEulerian triangulationが復元される。ルートノードを加える必要がある。三角形の復活に関して、塗り分け2色の片方であることから、探すべきnのノードはエッジの方向を考慮しながら、特定の領域にあるはずだ、という復元の仕方をしないといけないことに注意
- Eulerian TriangulationとWell-labeled treeを扱うにはPlanar graph特有の性質を使う必要がある。planar graph特有の性質とは、ノードに接続するエッジが周囲360度に関して順序があって、それを入れ替えたりできないこと。双対グラフを使うとそういう性質がハンドリングできるらしい→こちら
- まったくの書きかけだけれど…(そして実装することがゴールではなく、どういう性質でどういうアルゴリズムなのかがわかることが目的だったので、実装自体はちょっとめんどくさそうだし、書きかけのままにするだろうけれど…)
library(igraph)
n <- 10
edge.list <- matrix(c(1,2),1,2)
for(i in 3:n){
edge.list <- rbind(edge.list,c(sample(edge.list,1),i))
}
Rt <- n+1
edge.list.lb <- cbind(lb[edge.list[,1]],lb[edge.list[,2]])
edge.list.lb.sort <- t(apply(edge.list.lb,1,sort))
edge.list.lb.dir <- edge.list.lb[,1] < edge.list.lb[,2]
edge.list.sort <- edge.list
edge.list.sort[!edge.list.lb.dir,1] <- edge.list[!edge.list.lb.dir,2]
edge.list.sort[!edge.list.lb.dir,2] <- edge.list[!edge.list.lb.dir,1]
g <- graph.edgelist(edge.list)
plot(g)
ad.mat <- as.matrix(get.adjacency(g))
ad.mat2 <- ad.mat + (-1)*t(ad.mat)
rt <- sample(1:n,1)
lb <- shortest.paths(g,rt)+1
lb.2 <- c()
for(i in 1:n){
lb.2[i] <- paste(i,lb[i],sep="_")
}
plot(g,vertex.label=lb)
pl.layout <- layout.auto(g)
plot(g, layout=pl.layout , vertex.label=lb)
edge.order <- edge.order.2 <- list()
for(i in 1:n){
tmp <- neighbors(g,i,mode=3)
tmp2 <- t(matrix(pl.layout[tmp,],ncol=2))-pl.layout[i,]
angle <- Arg(tmp2[1,] + 1i*tmp2[2,])
tmp3 <- tmp[order(angle)]
edge.order[[i]] <- tmp[order(angle)]
edge.order.2[[i]] <- rep(0,length(edge.order[[i]]))
for(j in 1:length(edge.order[[i]])){
ttmp <- abs(edge.list[,1] - i)+abs(edge.list[,2]-edge.order[[i]][j])
if(length(which(ttmp==0))==1){
edge.order.2[[i]][j] <- which(ttmp==0)[1]
}else{
ttmp <- abs(edge.list[,2] - i)+abs(edge.list[,1]-edge.order[[i]][j])
edge.order.2[[i]][j] <- which(ttmp==0)[1]
}
}
}
plot(g, layout=pl.layout,vertex.label=lb.2)
target.st <- 1
target.edge <- which(edge.list.lb.sort[,1]==target.st)
target.edge.st <- edge.list.sort[target.edge,1]
target.edge.end <- edge.list.sort[target.edge,2]
new.edge <- rbind(edge.list,cbind(rep(Rt,2*length(target.edge)),c(target.edge.st,target.edge.end)))
new.lb <- c(lb,0)
g2 <- graph.edgelist(new.edge,directed=FALSE)
plot(g2,layout=rbind(pl.layout,rep(max(pl.layout),2)),vertex.label=new.lb)
plot(g2,layout=rbind(pl.layout,c(min(pl.layout),max(pl.layout))))
new.edge.order <- edge.order
for(i in 1:length(target.edge)){
tmp.st <- target.edge.st[i]
tmp.end <- target.edge.end[i]
tmp.id <- which(edge.order[[tmp.st]] == tmp.end)
tmp.len <- length(edge.order[[tmp.st]])
if(tmp.id==1){
pre <- c()
post <- edge.order[[tmp.st]][(tmp.id):tmp.len]
}else if (tmp.id==tmp.len){
pre <- edge.order[[tmp.st]][1:(tmp.id-1)]
post <- edge.order[[tmp.st]][(tmp.id):tmp.len]
}else{
pre <- edge.order[[tmp.st]][1:(tmp.id-1)]
post <- edge.order[[tmp.st]][(tmp.id):tmp.len]
}
new.edge.order[[tmp.st]] <- c(pre,Rt,post)
tmp.id <- which(edge.order[[tmp.end]] == tmp.st)
tmp.len <- length(edge.order[[tmp.end]])
if (tmp.id==tmp.len){
pre <- edge.order[[tmp.end]][1:(tmp.id)]
post <- c()
}else if(tmp.id==1){
pre <- edge.order[[tmp.end]][1:(tmp.id)]
post <- edge.order[[tmp.end]][(tmp.id+1):tmp.len]
}else{
pre <- edge.order[[tmp.end]][1:(tmp.id)]
post <- edge.order[[tmp.end]][(tmp.id+1):tmp.len]
}
new.edge.order[[tmp.end]] <- c(pre,Rt,post)
}
n.step <- max(lb)-1
for(i in 2:n.step){
target.st <- i
target.edge <- which(edge.list.lb.sort[,1]==target.st)
candidate.node <- which(lb == target.st-1)
target.edge.st <- edge.list.sort[target.edge,1]
target.edge.end <- edge.list.sort[target.edge,2]
for(j in 1:length(target.edge)){
tmp.path <- get.shortest.paths(g2,from=target.edge.end[j],t=candidate.node,output="both")
}
}
dis.eg <- get.shortest.paths(g,1,output="both")
と書いて、n個の四角形からできた平面グラフを表す)と木グラフとが一対一対応すること(Cori-Vauquelin-Schaeffer bijectionと呼ばれる関係)







