- ZDDは組み合わせの圧縮表現
- Simpathはグラフの経路全網羅のためのZDD構築のアルゴリズム(こちら)
- アルゴリズムの説明をしてもらったけれど、どうやるのだか分らなかったのでRで、べたべたに書いてみることにする
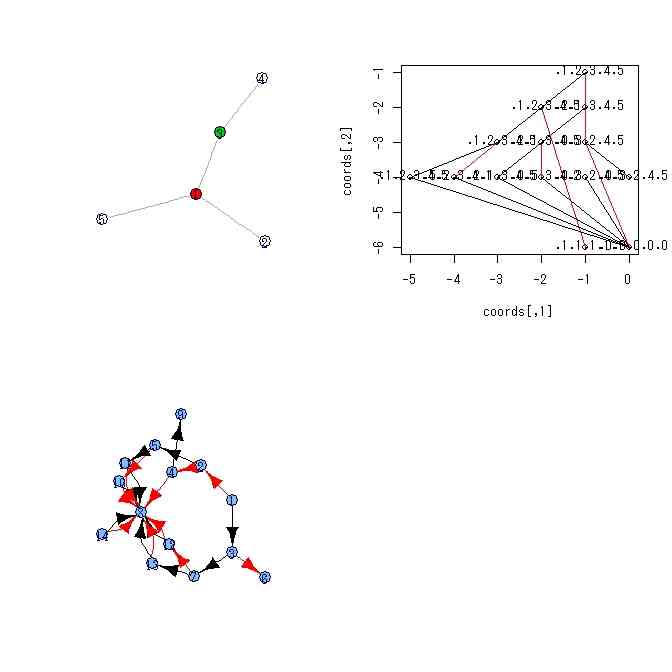
- グラフ
 がある
がある
 を定める
を定める とエッジに順序を定める
とエッジに順序を定める- 処理はすべてのエッジについてループを回す
- エッジを処理するごとにその処理段階ごとに「検討するべき状態」を決める
- 「検討するべき状態」についてループを回す
- 「検討するべき状態」からは、処理対象のエッジを加えるか加えないかの2つの「状態」が次の段階の「検討するべき状態」の候補として挙がる
- ただし、このままだと、「検討するべき状態」が倍々で増えてしまうので、以下をして「状態」を減らす
- (1)スタートからターゲットに到達したら、その状態については、以降の処理はしないので「検討するべき状態」には加えない
- (2)エッジを増やしても、経路として不適当(枝分かれ)だったら、以降の処理はしないので「検討するべき状態」には加えない
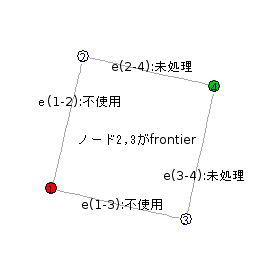
- (3)それ以外の場合は「mate」という条件に照らして、同じ場合は、「検討するべき状態」として同じなので、まとめる。ただし、考慮するべきは、線状になっている部分の両端で、かつ、それ以降に処理する予定のエッジがつながる可能性のあるノードについてのmate条件のみ。それをfrontierと言う。その説明の図を頂戴しました(下図)
- 全部のエッジの処理が済んだら、「最終的に経路になったもの」は「経路」とし、「最終的に経路とならなかったもの」は、「不採用」
- 「最後の不採用」の結果、ZDD的に省略できる枝は、刈り込む。刈り込むにあたって、処理した順番が後の方のエッジの方から刈り込んでいく(たぶんこれは最後にやるのだと思う…)
- これらを「きれいに」書くのが目標だが、「きれいに」書くと、後で読んでわからなくなるのが目に見えているので、べたべたに書く
- まずは、小さいグラフを作ろう(ここは大きすぎなければどうでもよい)
n.v <- 5
n.e <- sample((n.v+1):(n.v*(n.v-1)/2),1)
e.list <- cbind(sample(1:n.v,n.e,replace=TRUE),sample(1:n.v,n.e,replace=TRUE))
e.list <- e.list[which(e.list[,1]!=e.list[,2]),]
e.list
library(igraph)
g <- graph.edgelist(e.list,directed=FALSE)
m <- (sign(as.matrix(get.adjacency(g))))
m[upper.tri(m)] <- 0
g <- graph.adjacency(m,mode="undirected")
e.list <- get.edgelist(g)
goal <- sample(2:n.v,1)
goal
frontiers <- list()
for(i in 1:length(e.list[,1])){
frontiers[[i]] <- sort(unique(c(e.list[i:length(e.list[,1]),])))
}
P <- Pnew <- tmp.Pnew <- list()
fr.Pnew <- fr.tmp.Pnew <- list()
P[[1]] <- matrix(rep(1:n.v,2),nrow=2,byrow=TRUE)
ZDD.edge <- matrix(NA,0,5)
mates <- matrix(NA,0,n.v*2)
row.goal <- length(e.list[,1])+2
for(i in 1:length(e.list[,1])){
tmp.edge <- e.list[i,]
print("current edge is ")
print(tmp.edge)
id <- 0
if(length(P) > 0){
for(j in seq(P)){
print(P[[j]])
print("reached")
a <- P[[j]][,tmp.edge[1]]
b <- P[[j]][,tmp.edge[2]]
if(a[2]*b[2]==0){
print("branching")
ZDD.edge <- rbind(ZDD.edge,c(i,j,row.goal,0,1))
mates <- rbind(mates,c(P[[j]][2,],rep(0,n.v)))
}else{
print("add edge")
id <- length(Pnew)+1
tmp.Pnew[[1]] <- P[[j]]
x <- a[2]
y <- b[2]
z <- P[[j]][,x]
w <- P[[j]][,y]
print("z=")
print(z)
print("w=")
print(w)
tmp.Pnew[[1]][2,a[1]] <- tmp.Pnew[[1]][2,b[1]] <- 0
if(z[1]-z[2]!=0){
tmp.Pnew[[1]][2,z[1]] <- w[1]
}else{
tmp.Pnew[[1]][2,z[1]] <- w[1]
}
if(w[1]-w[2]!=0){
tmp.Pnew[[1]][2,w[1]] <- z[1]
}else{
tmp.Pnew[[1]][2,w[1]] <- z[1]
}
print(tmp.Pnew[[1]])
id.incl <- FALSE
if(tmp.Pnew[[1]][2,1] == goal){
print("goal")
ZDD.edge <- rbind(ZDD.edge,c(i,j,row.goal,1,1))
mates <- rbind(mates,c(P[[j]][2,],rep(1,n.v)))
}else if(length(which(tmp.Pnew[[1]][2,frontiers[[i]]]==1)) < 1){
ZDD.edge <- rbind(ZDD.edge,c(i,j,row.goal,0,1))
mates <- rbind(mates,c(P[[j]][2,],rep(0,n.v)))
}else{
if(i == length(e.list[,1])){
ZDD.edge <- rbind(ZDD.edge,c(i,j,row.goal,0,1))
mates <- rbind(mates,c(P[[j]][2,],rep(0,n.v)))
}else{
sameMate <- id
for(k in seq(Pnew)){
if(sum((Pnew[[k]][2,frontiers[[i]]]-tmp.Pnew[[1]][2,frontiers[[i]]])^2)==0){
sameMate <- k
}
}
if(sameMate == id){
Pnew[[length(Pnew)+1]] <- tmp.Pnew[[1]]
id.incl <- TRUE
}
ZDD.edge <- rbind(ZDD.edge,c(i,j,i+1,sameMate,1))
mates <- rbind(mates,c(P[[j]][2,],tmp.Pnew[[1]][2,]))
}
}
}
print("not add edge")
if(id.incl){
id <- id+1
}else{
id <- id
}
tmp.Pnew[[1]] <- P[[j]]
if(length(which(tmp.Pnew[[1]][2,frontiers[[i]]]==1)) < 1){
ZDD.edge <- rbind(ZDD.edge,c(i,j,row.goal,0,1))
mates <- rbind(mates,c(P[[j]][2,],rep(0,n.v)))
}else if(i == length(e.list[,1])){
ZDD.edge <- rbind(ZDD.edge,c(i,j,row.goal,0,0))
mates <- rbind(mates,c(P[[j]][2,],rep(0,n.v)))
}else{
sameMate <- id
for(k in seq(Pnew)){
if(sum((Pnew[[k]][2,frontiers[[i]]]-tmp.Pnew[[1]][2,frontiers[[i]]])^2)==0){
sameMate <- k
}
}
if(sameMate == id){
Pnew[[length(Pnew)+1]] <- tmp.Pnew[[1]]
}
ZDD.edge <- rbind(ZDD.edge,c(i,j,i+1,sameMate,0))
mates <- rbind(mates,c(P[[j]][2,],tmp.Pnew[[1]][2,]))
}
}
print("renew P")
P <- Pnew
Pnew <- list()
}
}
draw.dd <- function(dd){
nodes.1 <- paste(dd[,1],dd[,2],sep=".")
nodes.2 <- paste(dd[,3],dd[,4],sep=".")
e.list <- cbind(nodes.1,nodes.2)
g <- graph.edgelist(e.list)
E(g)$color <- dd[,5]+1
plot(g)
}
draw.dd.2 <- function(dd,mates){
dd.2 <- -dd
dd.2[,5] <- -dd.2[,5]
dd.2[,c(2,4)]
coords <- rbind(dd.2[,2:1],dd.2[,4:3])
plot(coords)
segments(dd.2[,2],dd.2[,1],dd.2[,4],dd.2[,3],col=dd.2[,5]+1)
labels <- rbind(mates[,1:n.v],mates[,(n.v+1):(n.v*2)])
labels2 <- rep(NULL,length(labels[,1]))
for(i in 1:length(labels[1,])){
labels2 <- paste(labels2,labels[,i],sep=".")
}
text(coords+0.1,labels2)
}
par(mfcol=c(2,2))
node.col <- rep(0,n.v)
node.col[1] <- 2
node.col[goal] <- 3
V(g)$color <- node.col
plot(g)
draw.dd(ZDD.edge)
draw.dd.2(ZDD.edge,mates)
e.list
がある
を定める
とエッジに順序を定める

