
- ユークリッド幾何は、長さが大事。平行線が交わらないことも大事
- 射影幾何は長さはどうでもよくなって、平行線は無限遠で交わり、すべての直線が交点を持つ
- 有限幾何が、点と線(と面と…)と組合せ論的関係性の抽象化したものであるように
- 射影幾何も「ユークリッド幾何」で慣れ親しんでいるもろもろの関係性を部分的に抽出したものになっている
- 射影幾何では円も楕円も双曲線も放物線も相互に移行し合える「同じもの〜二次曲線」となる。三角形は直角三角形も鋭角三角形も鈍角三角形も「三角形」として同じものになる
- 射影は透視絵と関係していて、あるd次元空間の点をある視点・光源から見て、d-1次元(超)平面に存在するものとする
- 射影変換は、d次元空間において、d-1次元(超)平面の点(の集合)を別のd-1次元(超)平面に射影写像して、今度は視点・光源をずらして、また、次のd-1次元(超)平面に移す。これを2回以上繰り返して、もとのd-1次元(超)平面に戻してやることができる。そうすると、もとのd-1次元(超)平面上の「点の集合(形に意味があったりする)」が、「別の点の集合」に移される。移されるけれど、「射影幾何的には同じもの〜円が楕円に変わっていたりするけれど、それが『違う』のはユークリッドな感覚の場合であって、射影幾何的には、『同じ』で、『射影幾何的「合同」変換」しただけです」と言う
- そんな射影変換で説明できる生物形態があるよ、という話
- まずは、射影写像をRでやろう
- 準備。任意次元空間で、法線ベクトルを指定して、(1,0,0,...)というベクトルをその法線ベクトルに回転して移す正規直交基底行列・回転行列を作れるようにしておく(こちらで作った『遺産』)
base.v <- function(v){
d <- length(v)
v <- v/sqrt(sum(v^2))
ret <- matrix(0,d,d)
for(i in 2:(d)){
ret[1:(i-1),i] <- v[1:(i-1)]
ret[i,i] <- -sum(v[1:(i-1)]^2)/v[i]
}
ret[,1] <- v
q <- sqrt(apply(ret^2,2,sum))
t(t(ret)/q)
}
out <- base.v(runif(4))
out %*% t(out)
my.projection <- function(x,L,p0,v){
if(!is.matrix(x)){
x <- matrix(x,ncol=1)
}
base1 <- base.v(v)
x. <- x-p0
L. <- L-p0
x.. <- t(base1) %*% x.
L.. <- t(base1) %*% L.
k <- -L..[1,]/(x..[1,]-L..[1,])
X <- matrix(0,length(L),length(x[1,]))
X <- c(L..) + t(k * t(x..-c(L..)))
X. <- base1 %*% X + p0
X.
}
v <- c(1,2)
p0 <- c(1,1)
L <- c(4,5)
t <- seq(from=0,to=1,length=100)
xs <- t*1
a <- 3
b <- 2
ys <- a*xs+b
XYs <- rbind(xs,ys)
p.XYs <- my.projection(XYs,L,p0,v)
xlim <- ylim <- range(c(c(p.XYs),c(XYs)))
plot(t(p.XYs),type="b",asp=TRUE,xlim=xlim,ylim=ylim)
points(t(XYs),col=2)
v <- runif(3)
p0 <- runif(3)
L <- runif(3)
library(MCMCpack)
r <- rdirichlet(1000,rep(0.4,3))
xvs <- matrix(rnorm(3*3),3,3)
Xs <- xvs %*% t(r)
p.Xs <- my.projection(Xs,L,p0,v)
library(rgl)
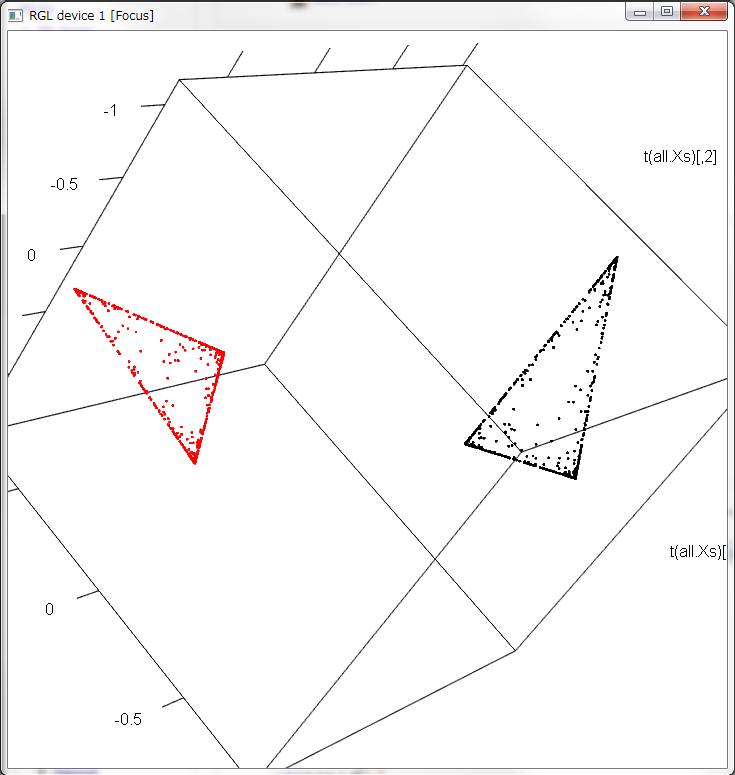
all.Xs <- cbind(Xs,p.Xs)
col <- rep(1:2,each=length(Xs[1,]))
plot3d(t(all.Xs),col=col)
plot3d(t(Xs))
plot3d(t(p.Xs))


