
- その中に、回転行列が回転群をなし、回転行列は四元数表示ができて、それらと被覆との関係の記事がある
- その応用例として人体骨格の回転行列表現があった
- 年明けの「バドミントン大会」に向けて、フォームの研究をしてみよう
- 骨格をグラフで表し
- エッジに回転行列を与えよう
- 回転行列はエッジをたどりながら、「累積」するので、そんな処理を入れる
- ひとまず、いろんな姿勢を取らせることだけを考えよう
- 後は、適当に各エッジの回転指定を変えて、取りたい姿勢を取るためにどんな回転指定をすればよいかを、トライアンドエラーで探索してみよう
- この段階は、「大脳皮質」による確認のフェーズ
- うまく行き始めたら、小脳的にスマートに動けるようになる(かもしれない)
n.node <- 16
body <- matrix(0,n.node,3)
edges <- matrix(0,n.node-1,2)
edges[1,] <- c(1,2)
edges[2,] <- c(2,3)
edges[3,] <- c(3,4)
edges[4,] <- c(3,5)
edges[5,] <- c(5,6)
edges[6,] <- c(6,7)
edges[7,] <- c(3,8)
edges[8,] <- c(8,9)
edges[9,] <- c(9,10)
edges[10,] <- c(1,11)
edges[11,] <- c(11,12)
edges[12,] <- c(12,13)
edges[13,] <- c(1,14)
edges[14,] <- c(14,15)
edges[15,] <- c(15,16)
library(igraph)
g <- graph.edgelist(edges,directed=FALSE)
sh.pt <- list()
sh.pt[[1]] <- c(1)
sh.pt[[2]] <- c(1,2)
sh.pt[[3]] <- c(1,2,3)
sh.pt[[4]] <- c(1,2,4)
sh.pt[[5]] <- c(sh.pt[[4]],5)
sh.pt[[6]] <- c(sh.pt[[5]],6)
sh.pt[[7]] <- c(sh.pt[[2]],7)
sh.pt[[8]] <- c(sh.pt[[7]],8)
sh.pt[[9]] <- c(sh.pt[[8]],9)
sh.pt[[10]] <- c(10)
sh.pt[[11]] <- c(10,11)
sh.pt[[12]] <- c(10,11,12)
sh.pt[[13]] <- c(13)
sh.pt[[14]] <- c(13,14)
sh.pt[[15]] <- c(13,14,15)
e.edge <- c(20,20,10,rep(c(10,25,25),2),rep(c(8,30,30),2))
e.v <- matrix(0,n.node-1,3)
e.v[1,] <- c(0,0,1)
e.v[2,] <- c(0,0,1)
e.v[3,] <- c(0,0,1)
e.v[4,] <- c(1,0,0)
e.v[5,] <- c(0,0,-1)
e.v[6,] <- c(0,0,-1)
e.v[7,] <- c(-1,0,0)
e.v[8:9,] <- e.v[5:6,]
e.v[10:12,] <- e.v[4:6,]
e.v[13:15,] <- e.v[7:9,]
R <- list()
for(i in 1:length(e.v[,1])){
R[[i]] <- diag(rep(1,3))
}
r <- rep(1,n.node)
r[4] <- 5
for(i in 1:length(edges[,1])){
body[edges[i,2],] <- body[edges[i,1],] + e.edge[i] * R[[i]] %*% e.v[i,]
}
library(rgl)
segs <- NULL
for(i in 1:length(edges[,1])){
segs <- rbind(segs, body[edges[i,1],])
segs <- rbind(segs, body[edges[i,2],])
}
max.body <- max(abs(body))
body.plus <- rbind(body,rep(max.body,3))
body.plus <- rbind(body.plus,rep(-max.body,3))
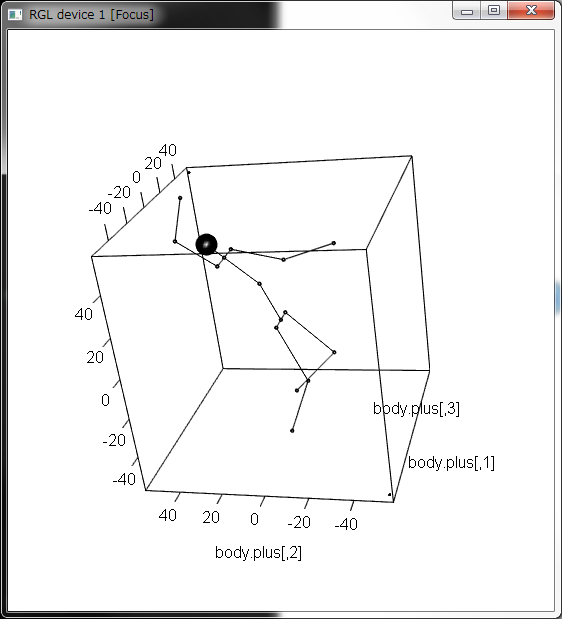
plot3d(body.plus)
segments3d(segs)
for(i in 1:n.node){
spheres3d(body[i,],radius = r[i])
}
make.rot <- function(v,theta){
v2 <- v/sqrt(sum(v^2))
ct <- cos(theta/2)
st <- sin(theta/2)
x <- st * v2[1]
y <- st * v2[2]
z <- st * v2[3]
w <- ct
matrix(c(1-2*y^2-2*z^2,2*x*y+2*w*z,2*x*z-2*w*y,2*x*y-2*w*z,1-2*x^2-2*z^2,2*y*z+2*w*x,2*x*z+2*w*y,2*y*z-2*w*x,1-2*x^2-2*y^2),byrow=TRUE,3,3)
}
make.rot(c(1,0,0),pi/2)
R <- list()
for(i in 1:length(e.v[,1])){
R[[i]] <- diag(rep(1,3))
}
R[[1]] <- make.rot(c(1,0,0),pi/8)
R[[2]] <- make.rot(c(1,0,0),pi/7)
R[[11]] <- make.rot(c(1,0,0),pi/4)
R[[12]] <- make.rot(c(1,0,0),-pi/2)
R[[14]] <- make.rot(c(1,0,0),pi/8)
R[[15]] <- make.rot(c(1,0,0),-pi/4)
R[[5]] <- make.rot(c(1,1,0),pi/6)
R[[6]] <- make.rot(c(1,-2,0),pi/2)
R[[8]] <- make.rot(c(1,-0.2,0),pi/0.9)
R[[9]] <- make.rot(c(-3,-2,0),pi/2)
calc.body <- function(body,edges,e.edge,e.v,sh.pt,R){
new.body <- body
new.R <- list()
for(i in 1:length(R)){
new.R[[i]] <- diag(rep(1,3))
for(j in 1:length(sh.pt[[i]])){
new.R[[i]] <- R[[sh.pt[[i]][j]]] %*% new.R[[i]]
}
}
for(i in 1:length(edges[,1])){
new.body[edges[i,2],] <- new.body[edges[i,1],] + e.edge[i] * new.R[[i]] %*% e.v[i,]
}
list(new.body = new.body, new.R = new.R)
}
body <- matrix(0,n.node,3)
calc.body.out <- calc.body(body,edges,e.edge,e.v,sh.pt,R)
body <- calc.body.out$new.body
library(rgl)
segs <- NULL
for(i in 1:length(edges[,1])){
segs <- rbind(segs, body[edges[i,1],])
segs <- rbind(segs, body[edges[i,2],])
}
max.body <- max(abs(body))
body.plus <- rbind(body,rep(max.body,3))
body.plus <- rbind(body.plus,rep(-max.body,3))
plot3d(body.plus)
segments3d(segs)
for(i in 1:n.node){
spheres3d(body[i,],radius = r[i])
}

