- 選択公理というのがある
- それに関連してバナッハ=タルスキーのパラドクスというのがある
- 球を2つに分ける話
- 分けるからには、ある点はどちらか片方の球にしか属していなくて
- 2つに分けたそれぞれは球である
- これは、バナッハ=タルスキーのパラドクスの本質的な部分を一部、欠いた説明とも言えるけれど、「この世」にバナッハ=タルスキーのパラドクスを応用しようとすれば、まあ、こんな感じで考えておいてもよいだろう
- 球にある回転を繰り返すこと
- 球を回転するとき、1回の回転で回る角度がちょうどよければ、何回か回転すると、ちょうど元に戻る
- 角度がちょうどよくないと、永遠に元に戻らない
- 角度の単位に
 という無理数があるので、有理数な角度の回転の繰り返しは「ちょうどよくない」方に属する
という無理数があるので、有理数な角度の回転の繰り返しは「ちょうどよくない」方に属する
- 球面は2次元なので、回転を組み合わせる
- 1種類の回転を続けると、ある点は、球面に1本の軌道を作り、それは円になる
- 球面は2次元なので、2つの異なる回転を「2次元酔歩」すると、球面を覆う酔歩軌道が描ける
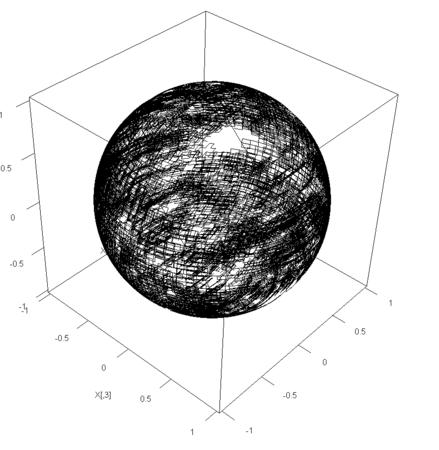
a <- 0.05
b <- 0.08
Ma <- matrix(c(cos(a),-sin(a),0,sin(a),cos(a),0,0,0,1),byrow=TRUE,3,3)
Mb <- matrix(c(cos(b),0,-sin(b),0,1,0,sin(b),0,cos(b)),byrow=TRUE,3,3)
Ms <- list()
Ms[[1]] <- Ma
Ms[[2]] <- t(Ma)
Ms[[3]] <- Mb
Ms[[4]] <- t(Mb)
s <- sample(1:4,100000,replace=TRUE)
s.2 <- c(s[1])
for(i in 2:length(s)){
tmp <- FALSE
if(s[i-1] ==1){
if(s[i] != 2){
tmp <- TRUE
}
}else if(s[i-1] == 2){
if(s[i] != 1){
tmp <- TRUE
}
}else if(s[i-1] ==3){
if(s[i] != 4){
tmp <- TRUE
}
}else{
if(s[i] != 3){
tmp <- TRUE
}
}
if(tmp) s.2 <- c(s.2,s[i])
}
length(s)-length(s.2)
s <- s.2
X <- matrix(0,length(s)+1,3)
X[1,] <- runif(3)
X[1,] <- X[1,]/sqrt(sum(X[1,]^2))
for(i in 2:length(X[,1])){
X[i,] <- Ms[[s[i-1]]] %*% X[i-1,]
}
library(rgl)
X <- rbind(X,diag(rep(1,3)),diag(rep(-1,3)))
plot3d(X,type="l")
- 2つに分けられることと自由群
- {a,-a,b,-b}が作るすべての配列であって、a -> (-a) という連続と、その逆の(-a) -> aという連続、同様に b -> (-b), (-b) -> bという連続は縮めて、除いてしまう、というルールで配列を作るとする
- a,-a,b,-bをそれぞれ回転として考えれば、正・逆の回転が続いたら、何もなかったことにしても同じだが、それに対応する手続きである
- このような配列を考える
ss <- list()
for(ii in 1:5){
s <- sample(c("a","-a","b","-b"),10,replace=TRUE)
s.2 <- c(s[1])
for(i in 2:length(s)){
if(s[i] != s.2[length(s.2)]) s.2 <- c(s.2,s[i])
}
length(s)-length(s.2)
s <- s.2
ss[[ii]] <-paste(s,collapse = ":")
}
ss
[[1]]
[1] "-b:-a:a:b:-a:b:-a"
[[2]]
[1] "a:-b:a:-b:b:-a:a"
[[3]]
[1] "-a:b:-b:a:-b:-a:a:b"
[[4]]
[1] "-a:-b:b:a:-a:-b:-a:b:-a"
[[5]]
[1] "-b:b:-a:b:-a:a:-a:b"
-
- これはまず、4種類に分けられる。それは、最初がa,-a,b,-bの4つのうちのいずれであるかでの分類である
- これらを
 と書くことにする
と書くことにする
- 絵を描こう
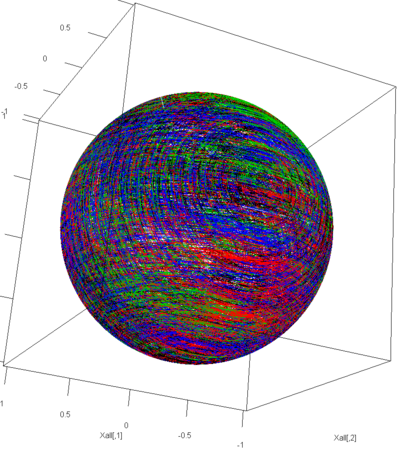
a <- 0.1
b <- 0.1
Ma <- matrix(c(cos(a),-sin(a),0,sin(a),cos(a),0,0,0,1),byrow=TRUE,3,3)
Mb <- matrix(c(cos(b),0,-sin(b),0,1,0,sin(b),0,cos(b)),byrow=TRUE,3,3)
Ms <- list()
Ms[[1]] <- Ma
Ms[[2]] <- t(Ma)
Ms[[3]] <- Mb
Ms[[4]] <- t(Mb)
X0 <- runif(3)
X0 <- X0/sqrt(sum(X0^2))
Xall <- matrix(c(1,1,1,-1,-1,-1),byrow=TRUE,nrow=2)
cols <- rep(0,2)
n.iter <- 12
n.step <- 10000
for(ii in 1:n.iter){
s <- sample(1:4,n.step,replace=TRUE)
s[1] <- ii %% 4 +1
tobeavoid <- c(2,1,4,3)
s[2] <- sample((1:4)[-tobeavoid[s[1]]],1)
s[3] <- sample((1:4)[-tobeavoid[s[2]]],1)
s.2 <- c(s[1])
for(i in 2:length(s)){
tmp <- FALSE
if(s[i-1] ==1){
if(s[i] != 2){
tmp <- TRUE
}
}else if(s[i-1] == 2){
if(s[i] != 1){
tmp <- TRUE
}
}else if(s[i-1] ==3){
if(s[i] != 4){
tmp <- TRUE
}
}else{
if(s[i] != 3){
tmp <- TRUE
}
}
if(tmp) s.2 <- c(s.2,s[i])
}
length(s)-length(s.2)
s <- s.2
X <- matrix(0,length(s)+1,3)
X[1,] <- X0
for(i in 2:length(X[,1])){
X[i,] <- Ms[[s[i-1]]] %*% X[i-1,]
}
Xall <- rbind(Xall,X)
cols <- c(cols,0,0,rep(s[1],length(s)+1-3),0)
}
plot3d(Xall,type="l",col=cols)
-
- この4種類を2種類に分ける方法として、最初がaかそれ以外か、というのもあるだろう
- この2分割が面白いのは、最初がaではない配列は
 と書けること(
と書けること( は初めが-aだが、それにaを付け加えると
は初めが-aだが、それにaを付け加えると の初めの-aが取れた配列が残るわけだが、
の初めの-aが取れた配列が残るわけだが、 の配列の2番目は、-a,b,-bのいずれかであって、aはないからそのようになる
の配列の2番目は、-a,b,-bのいずれかであって、aはないからそのようになる
- このことから、次のように言える
- ある球面上の1点から、ある2つの回転によって定義された折れ線的な軌道を考える
- 球面は軌道によって覆われる
- 特に、1ステップが結構な『歩幅』を持つとき、「ぐるりと回っても元の点に戻らない」という性質から、球面はかなりくまなく到達可能である
- さらに、その軌道は、第1歩が4種類のうちの一つに限定されるので、4種類のそれぞれが、球面をくまなく覆う
- この4種類は、始点の周辺では、明快に分かれているから、その根っこをぐっと握って、グイと引けば、球面全体をまんべんなく手繰り寄せることができる
- しかも、4種類のうち1種類を引っ張っても、行き着いている先を「うまく」とって来れば、球面の全体の半分にすることができそうである。「先の付着の仕組み」をうまいことしておけば、ですが(この「うまいこと」が選択公理的な「うまさ」だったり、「点」は、面積がないから、1:3の面積を比較しても、対等とみなすことができるというような「幾何学的なうまさ」だったり。
- 歩幅を粗大にした場合
a <- 1
b <- 1
Ma <- matrix(c(cos(a),-sin(a),0,sin(a),cos(a),0,0,0,1),byrow=TRUE,3,3)
Mb <- matrix(c(cos(b),0,-sin(b),0,1,0,sin(b),0,cos(b)),byrow=TRUE,3,3)
Ms <- list()
Ms[[1]] <- Ma
Ms[[2]] <- t(Ma)
Ms[[3]] <- Mb
Ms[[4]] <- t(Mb)
X0 <- runif(3)
X0 <- X0/sqrt(sum(X0^2))
Xall <- matrix(c(1,1,1,-1,-1,-1),byrow=TRUE,nrow=2)
cols <- rep(0,2)
n.iter <- 12
n.step <- 1000
for(ii in 1:n.iter){
s <- sample(1:4,n.step,replace=TRUE)
s[1] <- ii %% 4 +1
tobeavoid <- c(2,1,4,3)
s[2] <- sample((1:4)[-tobeavoid[s[1]]],1)
s[3] <- sample((1:4)[-tobeavoid[s[2]]],1)
s.2 <- c(s[1])
for(i in 2:length(s)){
tmp <- FALSE
if(s[i-1] ==1){
if(s[i] != 2){
tmp <- TRUE
}
}else if(s[i-1] == 2){
if(s[i] != 1){
tmp <- TRUE
}
}else if(s[i-1] ==3){
if(s[i] != 4){
tmp <- TRUE
}
}else{
if(s[i] != 3){
tmp <- TRUE
}
}
if(tmp) s.2 <- c(s.2,s[i])
}
length(s)-length(s.2)
s <- s.2
X <- matrix(0,length(s)+1,3)
X[1,] <- X0
for(i in 2:length(X[,1])){
X[i,] <- Ms[[s[i-1]]] %*% X[i-1,]
}
Xall <- rbind(Xall,X)
cols <- c(cols,0,0,rep(s[1],length(s)+1-3),0)
}
plot3d(Xall,type="l",col=cols)

n.t <- 20
kizami <- 0.1
group1 <- which(cols==2)
maxcoord <- n.t * kizami
for(i in 1:n.t){
tmpX <- Xall
tmpX2 <- Xall[group1,]
tmpX2[,1] <- tmpX2[,1]+0.1*i
tmpX[group1,] <- tmpX2
tmpX <- rbind(tmpX,rep(maxcoord+1,3),rep(-maxcoord,3))
plot3d(tmpX,type="l",col=c(cols,rep(0,2)))
Sys.sleep(10)
}
for(i in n.t:0){
tmpX <- Xall
tmpX2 <- Xall[group1,]
tmpX2[,1] <- tmpX2[,1]+0.1*i
tmpX[group1,] <- tmpX2
tmpX <- rbind(tmpX,rep(maxcoord+1,3),rep(-maxcoord,3))
plot3d(tmpX,type="l",col=c(cols,rep(0,2)))
open3d()
}
library(animation)
saveGIF({
for(i in 1:n.t){
tmpX <- Xall
tmpX2 <- Xall[group1,]
tmpX2[,1] <- tmpX2[,1]+0.1*i
tmpX[group1,] <- tmpX2
tmpX <- rbind(tmpX,rep(maxcoord+1,3),rep(-maxcoord,3))
plot3d(tmpX,type="l",col=c(cols,rep(0,2)))
}
},interval=0.05)
- さらに…
- さっさーと動ける要素(切ってはつなぐトポイソメラーゼのようなものがあれば…)もあれば、「滓(おり)」のように残ってしまうものもあるとすると…細胞分裂のcontractile ringのようなものも見えてくる
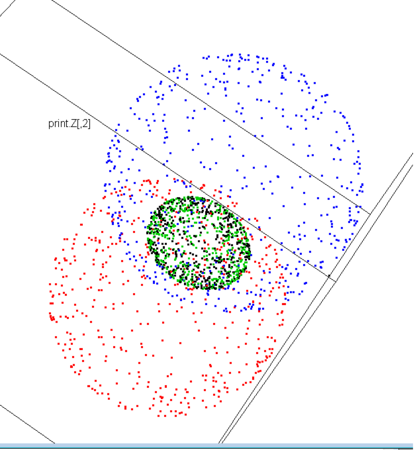
n.pt <- 1000
X1 <- matrix(rnorm(n.pt*3),ncol=3)
X2 <- matrix(rnorm(n.pt*3),ncol=3)
cols <- c(rep(1,n.pt/2),rep(2,n.pt/2),rep(3,n.pt/2),rep(4,n.pt/2))
Y <- rbind(X1,X2)
Y <- Y/sqrt(apply(Y^2,1,sum))
plot3d(Y,col=cols)
n.t <- 25
kizami <- 0.1
for(i in 0:n.t){
center1 <- 0
center2 <- i*kizami
tmp <- Y[1:n.pt,]
tmp[,1] <- tmp[,1] + kizami*i
Z <- Y
Z[1:n.pt,] <- tmp
s1 <- 1:(n.pt/2)
s2 <- (n.pt+1):(n.pt*3/2)
tmp1 <- Z[s1,]
tmp2 <- Z[s2,]
tmp3 <- tmp2
tmp3[,1] <- tmp3[,1]-i*kizami
t1 <- which(sqrt(apply(tmp1^2,1,sum))>1)
t2 <- which(sqrt(apply(tmp3^2,1,sum))>1)
x <- i*kizami/2
r <- sqrt(1-x^2)
tmp1.1 <- tmp1[t1,]
tmp1.1[,1] <- x
tmp1.1[,2] <- tmp1.1[,2]*r
tmp1.1[,3] <- tmp1.1[,3]*r
tmp1[t1,] <- tmp1.1
tmp2.1 <- tmp2[t2,]
tmp2.1[,1] <- x
tmp2.1[,2] <- tmp2.1[,2]*r
tmp2.1[,3] <- tmp2.1[,3]*r
tmp2[t2,] <- tmp2.1
Z[s1,] <- tmp1
Z[s2,] <- tmp2
print.Z <- rbind(Z,c(3,3,3),c(-1,-1,-1))
plot3d(print.Z,col=cols)
Sys.sleep(0.5)
}
- 繰り返せるように
- 上記のようにしてできた2つの球は、同じ方式でさらに分裂して行ってほしい
- そのときに気になるのは、軌道のセットが、分裂後も分裂前と同様に4方向にバラバラになっていること
- これは、引っ張るときに使った「根」の部分と、それに続くもう1セグメントを破壊して引き寄せることで達成可能
- また、上記のような球の分裂は、あっち方向にでこっち方向にも起こしたい。それにはすでに対応している
- 均質性と不均質性
- 上記では、均質な2つの球への分裂が実現できた
- 不均質な分裂も可能だろうか
- 軌道メッシュの長さが不十分だと、分裂の際に、近傍を中心に引き抜かれる傾向が出る
- この性質は、極性を持った分裂を表しているかもしれない
- さらに言えば、メッシュの根っこが少しずつ破壊しながら分裂しているとき、特に分裂頻度が高いとき、メッシュ軌道は徐々に短くなり、特段の仕組みを入れなくても、だんだんに極性を伴った分裂が起きてくることになる
- 完全再生
- 上の話は、実は、「パラドクス」ではなくて、ただの「(均等)分割」の話
- 本当は「2つの完全な球」を作らなくていけない
 をまず
をまず と
と との2つに分ける
との2つに分ける に
に とすると、
とすると、 となるから、これと
となるから、これと とを合わせると
とを合わせると との完全なものができる
との完全なものができる- 同様に
 からも
からも が作れる
が作れる
- これで「完全な2つの球」にできた(こちらを参照)
 と
と とに分けるというのは、平面4方向を縦と横とに分けること
とに分けるというのは、平面4方向を縦と横とに分けること- これは中心小体のL字構造(こちらやこちら)と相照らし合っているのでは…(細胞分裂というより染色体分配だけれども)
- では、この分裂後の変換
 をやってみよう
をやってみよう
- また、分裂後に
 と
と をやる、というのも、ありそうな手である。どちらかに限定せずに、最初の回転を除去する、という「全部一斉に」という処理だから
をやる、というのも、ありそうな手である。どちらかに限定せずに、最初の回転を除去する、という「全部一斉に」という処理だから
- 後者のやり方の方が、「有限」の軌道の場合には、完全な再生に近い
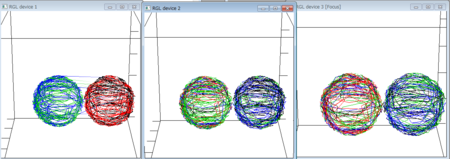
- 図は、左が、分離直後、中央がその後、
 のみを実施したもの、右は、
のみを実施したもの、右は、 を実施したもの
を実施したもの
library(rgl)
a <- 1
b <- 1
Ma <- matrix(c(cos(a),-sin(a),0,sin(a),cos(a),0,0,0,1),byrow=TRUE,3,3)
Mb <- matrix(c(cos(b),0,-sin(b),0,1,0,sin(b),0,cos(b)),byrow=TRUE,3,3)
Ms <- list()
Ms[[1]] <- Ma
Ms[[2]] <- t(Ma)
Ms[[3]] <- Mb
Ms[[4]] <- t(Mb)
X0 <- runif(3)
X0 <- X0/sqrt(sum(X0^2))
Xall <- matrix(c(1,1,1,-1,-1,-1),byrow=TRUE,nrow=2)
s.list <- list()
cols <- rep(0,2)
n.iter <- 12
n.step <- 100
for(ii in 1:n.iter){
s <- sample(1:4,n.step,replace=TRUE)
s[1] <- ii %% 4 +1
tobeavoid <- c(2,1,4,3)
s[2] <- sample((1:4)[-tobeavoid[s[1]]],1)
s[3] <- sample((1:4)[-tobeavoid[s[2]]],1)
s.2 <- c(s[1])
for(i in 2:length(s)){
tmp <- FALSE
if(s[i-1] ==1){
if(s[i] != 2){
tmp <- TRUE
}
}else if(s[i-1] == 2){
if(s[i] != 1){
tmp <- TRUE
}
}else if(s[i-1] ==3){
if(s[i] != 4){
tmp <- TRUE
}
}else{
if(s[i] != 3){
tmp <- TRUE
}
}
if(tmp) s.2 <- c(s.2,s[i])
}
length(s)-length(s.2)
s <- s.2
s.list[[ii]] <- s
X <- matrix(0,length(s)+1,3)
X[1,] <- X0
for(i in 2:length(X[,1])){
X[i,] <- Ms[[s[i-1]]] %*% X[i-1,]
}
Xall <- rbind(Xall,X)
cols <- c(cols,0,0,rep(s[1],length(s)+1-3),0)
}
plot3d(Xall,type="l",col=cols)
n.t <- 20
kizami <- 0.1
group1 <- which(cols<=2)
maxcoord <- n.t * kizami
for(i in 1:n.t){
tmpX <- Xall
tmpX2 <- Xall[group1,]
tmpX2[,1] <- tmpX2[,1]+kizami*i
tmpX[group1,] <- tmpX2
tmpX <- rbind(tmpX,rep(maxcoord+1,3),rep(-maxcoord,3))
plot3d(tmpX,type="l",col=c(cols,rep(0,2)))
Sys.sleep(0.5)
}
s.list.new <- s.list
for(i in 1:length(s.list.new)){
if(s.list[[i]][1] ==2) s.list.new[[i]] <- s.list[[i]][-1]
if(s.list[[i]][1] ==4) s.list.new[[i]] <- s.list[[i]][-1]
}
Xall.new <- matrix(c(1,1,1,-1,-1,-1),byrow=TRUE,nrow=2)
cols.new <- rep(0,2)
for(ii in 1:length(s.list.new)){
X <- matrix(0,length(s.list.new[[ii]])+1,3)
X[1,] <- X0
for(i in 2:length(X[,1])){
X[i,] <- Ms[[s.list.new[[ii]][i-1]]] %*% X[i-1,]
}
if(s.list[[ii]][1] <=2){
X[,1] <- X[,1] + kizami * n.t
}
Xall.new <- rbind(Xall.new,X)
cols.new <- c(cols.new,0,0,rep(s.list.new[[ii]][1],length(s.list.new[[ii]])+1-3),0)
}
open3d()
tmpX.all <- rbind(Xall.new,rep(maxcoord+1,3),rep(-maxcoord,3))
plot3d(tmpX.all,type="l",col = cols.new)
s.list.new <- s.list
for(i in 1:length(s.list.new)){
s.list.new[[i]] <- s.list[[i]][-1]
}
Xall.new <- matrix(c(1,1,1,-1,-1,-1),byrow=TRUE,nrow=2)
cols.new <- rep(0,2)
for(ii in 1:length(s.list.new)){
X <- matrix(0,length(s.list.new[[ii]])+1,3)
X[1,] <- X0
for(i in 2:length(X[,1])){
X[i,] <- Ms[[s.list.new[[ii]][i-1]]] %*% X[i-1,]
}
if(s.list[[ii]][1] <=2){
X[,1] <- X[,1] + kizami * n.t
}
Xall.new <- rbind(Xall.new,X)
cols.new <- c(cols.new,0,0,rep(s.list.new[[ii]][1],length(s.list.new[[ii]])+1-3),0)
}
open3d()
tmpX.all <- rbind(Xall.new,rep(maxcoord+1,3),rep(-maxcoord,3))
plot3d(tmpX.all,type="l",col = cols.new)
と書くことにする
と書けること(
は初めが-aだが、それにaを付け加えると
の初めの-aが取れた配列が残るわけだが、
の配列の2番目は、-a,b,-bのいずれかであって、aはないからそのようになる
をまず
と
との2つに分ける
に
とすると、
となるから、これと
とを合わせると
との完全なものができる
からも
が作れる
と
とに分けるというのは、平面4方向を縦と横とに分けること
をやってみよう
と
をやる、というのも、ありそうな手である。どちらかに限定せずに、最初の回転を除去する、という「全部一斉に」という処理だから
のみを実施したもの、右は、
を実施したもの





